Los microbiomas del intestino de los peces consisten de miles de taxones de bacterias que interactúan entre ellas, su ambiente y el huésped; y están sometidas a la complejidad y fluctuaciones dinámicas, y su entorno, estilo de vida y dieta.
Por otro lado, la microbiota intestinal en los peces es un criterio fiable para evaluar el éxito de la cría selectiva con cambios en la composición de la dieta, en especies como por ejemplo la dorada (Sparus aurata).
Sin embargo, el conocimiento de la dinámica en la microbiota intestinal en los peces aún se encuentra en estado inicial, debido a su complejidad, los múltiples factores bióticos y abióticos involucrados.
En este sentido, los investigadores del Institute of Aquaculture Torre de la Sal (IATS), Biotechvana, de la Universitat de Valencia, del Health Research Institute INCLIVA, del Principe Felipe Research Center (CIPF) y de la Foundation for the Promotion of Sanitary and Biomedical Research of the Valencian Community (FISABIO) desarrollaron un herramienta computacional que usa un enfoque de red bayesiano unificado para modelar la estructura de red de microbiomas intestinales de peces y sus interacciones con variables bióticas y abióticas asociadas con sistemas acuícolas típicos.
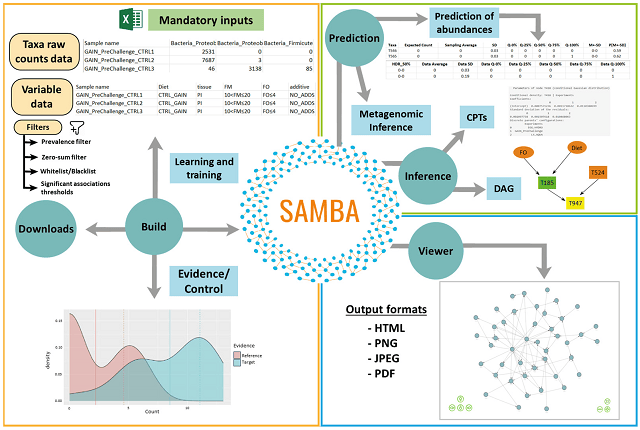
La herramienta denominada SAMBA (Structure-Learning of Aquaculture Microbiomes using a Bayesian Approach) acepta datos de entrada sobre la abundancia microbiana de amplicones de ARNr 16S, así como información continua y categórica de distintas condiciones de cultivo.
SAMBA puede crear y entrenar un escenario de modelo de red que se puede usar para:
- Inferir información sobre cómo las condiciones específicas de cultivo influyen en la diversidad del microbioma intestinal o pan-microbioma, y
- Predecir cómo la diversidad y el perfil funcional de ese microbioma cambiaría bajo otras condiciones variables.
El estudio presenta ejemplos y resultado de pruebas de escenarios de redes bayesianas creados por SAMBA utilizando datos de una comunidad microbiana sintética y el pan-microbioma de la dorada (Sparus aurata) en diferentes ensayos de alimentación.
Módulos de SAMBA
La herramienta SAMBA se encuentra disponible en un repositorio público GitHub, y puede ser instalada en las computadoras personales.
El componente frontal de SAMBA consiste en una interfaz gráfica de usuario (GUI) basada en la web implementada usando shiny para proporcionar una interfaz amigable.
Según el estudio, las funciones y tareas de SAMBA se estructuran en cinco módulos: “Construir”, “Inferencia”, “Predicción”, “Visor” y “Descargas”. El estudio incluye un archivo complementario donde puedes acceder a una guía del usuario con detalles técnicos.
Modelamiento de la microbiota
“Modelar las relaciones complejas de los componentes físicos y biológicos es particularmente relevante para la piscicultura porque la dinámica y las jerarquías de los microbiomas y pan-microbiomas de los peces pueden revelar información sobre los efectos de la genética, el medio ambiente o los factores de trazabilidad” destacaron los investigadores.
El software SAMBA se puede reutilizar para investigar las relaciones causales entre los microbiomas y sus anfitriones al descifrar cómo la poblaciones de taxones se relaciona entre sí e influye en las variables experimentales.
Además, reportan los investigadores: “las dependencias condicionales de BN de SAMBA brindan una perspectiva de biología del sistema que, en comparación con los análisis convencionales basados en la abundancia relativa de los grupos taxonómicos, es altamente informativo porque el usuario puede encontrar acciones combinadas entre taxones sin hacer suposiciones de independencia, como normalmente se realiza en un análisis de conteo 16S habitual”.
Potencial predictivo
Para probar el potencial y el poder predictivo de SAMBA, los investigadores “alimentaron” a la herramienta con dos grupos de datos de entrenamiento: una comunidad mock y la base de datos de S. aurata, para crear dos modelos de redes Bayesianas.
“Podemos concluir que las predicciones de SAMBA se aproximan con precisión a la observación del mundo real a partir de escenarios microbianos simplificados hechos en laboratorio, lo que podría ser particularmente útil para diseñar y explorar experimentos de biología sintética”, destacaron.
Según los investigadores SAMBA es una herramienta útil, que permite al usuario avanzar en la comprensión de la dinámica de la comunidad microbiana.
“La identificación de qué bacterias son más susceptibles de cambiar en una dieta y la magnitud en que cambian su abundancia, representa un pilar para la nutrición en la acuicultura”, informaron.
Los resultados del estudio destacan la capacidad de SAMBA para identificar los niveles de la dieta de harina y aceite de pescado que tienen una influencia significativa del perfil de la microbiota de los peces de cultivo.
El estudio fue financiado por el proyecto MCIN Bream-AquaINTECH para JP-S. El estudio también forma parte del programa ThinkInAzul.
Referencia (acceso abierto)
Soriano, B.; Hafez, A.I.; Naya-Català, F.; Moroni, F.; Moldovan, R.A.; Toxqui-Rodríguez, S.; Piazzon, M.C.; Arnau, V.; Llorens, C.; Pérez-Sánchez, J. SAMBA: Structure-Learning of Aquaculture Microbiomes Using a Bayesian Approach. Genes 2023, 14, 1650. https://doi.org/10.3390/genes14081650
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.

