La detección oportuna de patógenos persistentes y emergentes es fundamental para controlar la propagación de enfermedades, particularmente en poblaciones de alta densidad de camarones.
Las pruebas estándar de diagnóstico molecular para estudiar los microbios patógenos han brindado la sensibilidad necesaria para la detección temprana, pero el retraso en el tiempo de obtención de resultados conduce a una acción retardada.
Los diagnósticos in situ alivian el retraso, pero las tecnologías actuales son menos sensibles y adaptables que los métodos moleculares de laboratorio.
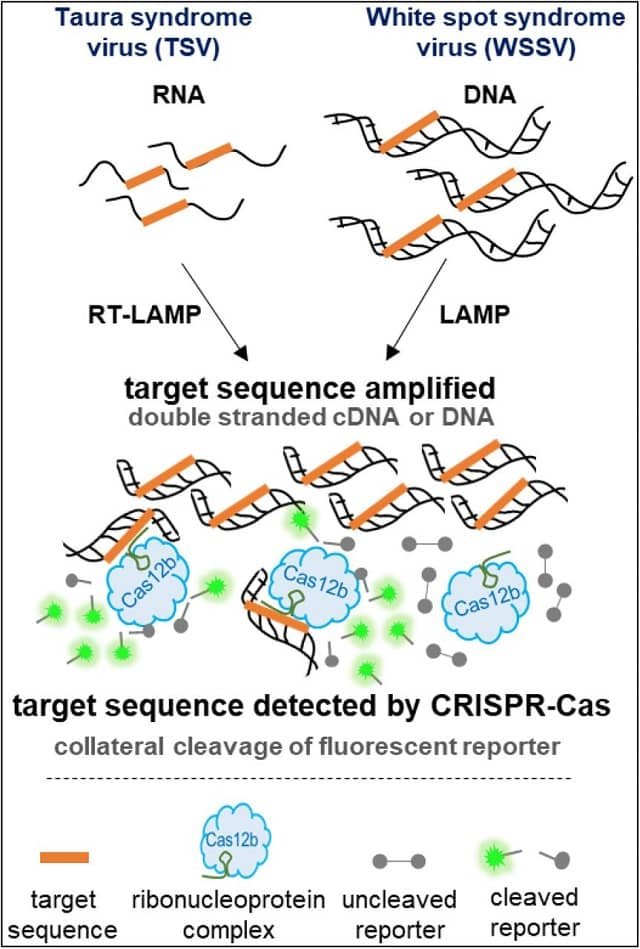
En este contexto, un equipo de investigadores de Gloucester Marine Genomics Institute, del Aquaculture Pathology Laboratory de la University of Arizona, desarrollaron el ensayo de diagnóstico SHERLOCKv2 basado en CRISPR para detectar virus de ADN y ARN que afectan al camarón, y presentan ensayos de detección del virus del Síndrome de la Mancha Blanca (WSSV) y del virus del Síndrome de Taura (TSV).
Desarrollo de ensayo de diagnóstico
Los investigadores combinaron la solidez de LAMP con la especificidad de CRISPR y la detección de la actividad de escisión colateral de Cas12b, para desarrollar ensayos SHERLOCKv2 de un solo recipiente sensibles y específicos para la detección de virus de ADN y ARN que afectan a los camarones.
En un etapa preliminar identificaron un conjunto de cebadores LAMP óptimo dirigido a una región del genoma de TSV, y lo compararon con la actividad de amplificación de tres conjuntos de cebadores, así como con un conjunto de cebadores LAMP publicados previamente por el mismo equipo de investigadores.
“Descubrimos que el conjunto publicado anteriormente dirigido al gen de la proteína de la cápside entre los dominios VP1 y VP3 mostró la detección más rápida y sensible de TSV y seleccionamos este conjunto para el ensayo SHERLOCKv2”, describieron.
Asimismo, reportan que al diseñar el ensayo WSSV, la proteína viral altamente conservada 28 (VP28) se centró en limitar la capacidad de la cepa WSSV para influir en la detección.
Validación del ensayo
Para estimar la sensibilidad del diagnóstico de los ensayos SHERLOCKv2, los investigadores evaluaron muestras de diferentes experimentos de desafío de virus del Síndrome del Taura y Virus del Síndrome de la Mancha Blanca en ensayos SHERLOCKv2 y en el diagnóstico de PCR para TSV y WSSV en tiempo real recomendado por la Organización Mundial de Sanidad Animal (OIE).
“Los ensayos se realizaron junto con curvas estándar para cuantificar y comparar el número de copias virales determinado por cada método”, reportan.
Asimismo, en los experimentos de desafío con TSV, debido a que la mayoría de las líneas genéticas de camarones disponibles comercialmente son tolerantes al TSV, los investigadores emplearon dos líneas comerciales diferentes de Penaeus vannamei que se conoce que difieren en su susceptibilidad.
El ensayo TSV SHERLOCK detectó TSV en 26 de las 30 muestras de camarones desafiadas con TSV.
“Para WSSV, evaluamos las mismas muestras de P. vannamei infectados con WSSV previamente cuantificados por qPCR recomendado por la OIE y el ensayo SHERLOCKv1”, dijeron.
Principales resultados
Según los investigadores, los ensayos fluorescentes pueden detectar el objetivo viral en el tejido del camarón en menos de una hora, y pueden adaptarse rápidamente a condiciones de campo para su uso en las granjas camaroneras.
“Ambos ensayos fluorescentes basados en CRISPR que desarrollamos mostraron una sensibilidad y precisión similares para la detección viral y la cuantificación de la carga para la PCR en tiempo real”, reportan los investigadores.
Asimismo destacan que ambos ensayos se dirigieron específicamente a sus respectivos virus sin detectar falsos positivos en animales infectados con otros patógenos comunes o en animales certificados como libres de patógenos específicos.
Conclusión
“Con una necesidad creciente de apoyar la producción acuícola, demostramos la capacidad de adaptar un ensayo SHERLOCK desarrollado para diagnóstico humano para detectar patógenos de ARN y ADN que afectan al camarón”, indican los investigadores.
“Esta demostración de la adaptación del ensayo para diversas enfermedades del camarón allana el camino para el desarrollo de ensayos de próxima generación en patología veterinaria y diagnóstico animal para mejorar el manejo de enfermedades en la acuicultura”, concluyen.
Referencia (acceso abierto)
Samuel R. Major, Matthew J. Harke, Roberto Cruz-Flores, Arun K. Dhar, Andrea G. Bodnar, Shelly A. Trigg. 2022. Rapid detection of DNA and RNA shrimp viruses using CRISPR-based diagnostics. bioRxiv 2022.12.14.520450; doi: https://doi.org/10.1101/2022.12.14.520450
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.

