El camarón blanco del Pacífico, Penaeus vannamei, ha revolucionado la industria acuícola mundial. Domesticada alrededor de 1980, esta especie se ha convertido en el crustáceo más cultivado. Sin embargo, las implicaciones genéticas de esta rápida domesticación son un tema de interés científico continuo.
Para comprender mejor la diversidad genética de esta especie, los científicos de CSIRO Agriculture and Food y de Genics analizaron 44.522 polimorfismos de un solo nucleótido (SNP) de 21 poblaciones cultivadas distribuidas globalmente y 86 P. vannamei silvestres. Los resultados brindan información valiosa sobre la composición genética de esta importante especie de acuicultura.
- 1 Una historia compleja de domesticación
- 2 Desafíos en la gestión de la diversidad genética
- 3 Avances recientes en herramientas de genotipado
- 4 Las poblaciones silvestres muestran una estructura poblacional débil
- 5 Las poblaciones de cultivo muestran niveles bajos a moderados de diferenciación poblacional
- 6 La secuenciación del genoma completo revela una diversidad de nucleótidos similar
- 7 Los barridos de selección identifican regiones y genes atípicos
- 8 Conclusión
- 9 Entradas relacionadas:
Una historia compleja de domesticación
La domesticación de P. vannamei es un esfuerzo relativamente reciente, que se remonta a fines de la década de 1980. Este proceso implicó la obtención de individuos silvestres del Océano Pacífico oriental, principalmente desde México hasta el norte de Perú.
Los primeros esfuerzos de domesticación se centraron en mejorar las tasas de crecimiento y la resistencia a las enfermedades mediante la cría selectiva. A medida que la industria se expandió, también lo hizo la diversidad genética de las poblaciones cultivadas, y el material genético se intercambió entre diferentes países y regiones.
Desafíos en la gestión de la diversidad genética
Uno de los desafíos clave en la gestión de la diversidad genética en las poblaciones de reproductores de P. vannamei es el riesgo de endogamia y reducción de la aptitud biológica debido a la alta fecundidad de la especie. Las herramientas de genotipado que informan los niveles de diversidad genética y respaldan estrategias de mejoramiento más avanzadas son esenciales para sustentar la ganancia genética sostenida y el crecimiento de la industria.
Avances recientes en herramientas de genotipado
Los avances recientes en herramientas de genotipado han permitido el desarrollo de dos mapas de ligamiento basados en SNP y una matriz de genotipado de 7 K SNP para P. vannamei. Estas herramientas han allanado el camino para los estudios de fenotipo a genotipo y han permitido la evaluación de la diversidad genética en poblaciones de cría comercial.
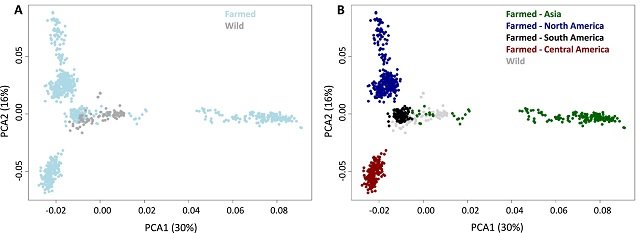
Las poblaciones silvestres muestran una estructura poblacional débil
El estudio encontró que las poblaciones silvestres de Panamá, Honduras, Guatemala y Colombia exhibieron una estructura poblacional débil, con una diferenciación genética de solo 0-3%. En contraste, se encontró que las poblaciones silvestres de México eran más distintas, lo que sugiere que pueden representar una subpoblación separada. Esta falta de una estructura poblacional fuerte entre las poblaciones silvestres puede deberse a la capacidad de la especie para dispersarse y migrar a través de grandes distancias.
Las poblaciones de cultivo muestran niveles bajos a moderados de diferenciación poblacional
Al comparar las poblaciones de cultivo y las silvestres, el estudio encontró niveles bajos a moderados de diferenciación poblacional, siendo las poblaciones asiáticas de cultivo las más divergentes. Esto sugiere que, si bien ha habido cierta deriva genética y selección en las poblaciones de cultivo, se ha mantenido la diversidad genética general.
La secuenciación del genoma completo revela una diversidad de nucleótidos similar
Para garantizar que los resultados no estuvieran influenciados por el sesgo de verificación, el estudio utilizó la secuenciación del genoma completo para identificar 4,13 millones de SNP en un subconjunto de camarones de cultivo y silvestres.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Los resultados mostraron que los niveles observados de diversidad de nucleótidos en animales silvestres y de cultivo eran similares, lo que sugiere que el proceso de domesticación no ha llevado a una reducción significativa de la diversidad genética. Es probable que esto se deba a la introducción repetida de camarones silvestres en poblaciones cultivadas, lo que ha ayudado a mantener la diversidad genética de la especie.
Los barridos de selección identifican regiones y genes atípicos
El estudio también identificó una pequeña colección de regiones atípicas y 20 genes codificadores de proteínas que pueden haber sido objeto de barridos de selección. Estas regiones y genes pueden haber sido favorecidos por la selección artificial en poblaciones cultivadas, lo que llevó a cambios en el fenotipo de la especie.
Conclusión
El estudio proporciona una instantánea completa de la diversidad genética y la divergencia dentro del camarón blanco del Pacífico P. vannamei. Los resultados del estudio se pueden resumir en:
- La domesticación ha dado lugar a agrupaciones genéticas distintas: las poblaciones de camarones cultivados de diferentes regiones geográficas han experimentado cambios significativos en las frecuencias de alelos desde que comenzó la domesticación en la década de 1980, lo que ha dado lugar a agrupaciones distintas.
- Se ha perdido poca diversidad genética en las poblaciones cultivadas: a pesar de la domesticación y la selección para el rendimiento, la diversidad genética del camarón cultivado se ha mantenido relativamente alta, con poca pérdida de diversidad en comparación con las poblaciones silvestres.
- El camarón silvestre tiene características genéticas únicas: los datos de secuenciación del genoma completo revelaron que el camarón silvestre tiene un exceso de alelos privados, que no se encuentran en las poblaciones cultivadas. Esto sugiere que el camarón silvestre tiene una composición genética única que podría ser beneficiosa para la acuicultura.
- La introducción de reproductores silvestres puede complementar la diversidad genética: el estudio sugiere que la incorporación de reproductores capturados en estado silvestre a las poblaciones de cultivo podría ayudar a complementar la diversidad genética, en particular en áreas donde se ha reducido.
En términos prácticos, estos hallazgos tienen implicaciones importantes para la gestión y conservación de las poblaciones de P. vannamei. Al incorporar reproductores silvestres a las poblaciones de cultivo, las industrias de la acuicultura pueden ayudar a mantener la diversidad genética y potencialmente mejorar la resiliencia y adaptabilidad de sus poblaciones de camarones.
Contacto
James Kijas
CSIRO Agriculture and Food
Brisbane, Queensland 4067, Australia
Email: james.kijas@csiro.au
Referencia (acceso abierto)
Kijas, J., Carvalheiro, R., Menzies, M., McWilliam, S., Coman, G., Foote, A., Moser, R., Franz, L., & Sellars, M. (2025). Genome-wide SNP variation reveals genetic structure and high levels of diversity in a global survey of wild and farmed Pacific white shrimp. Aquaculture, 597, 741911. https://doi.org/10.1016/j.aquaculture.2024.741911
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.