Proyecto Aquaimpact presenta resultados de que la selección genómica se puede implementar con precisión con un número mucho menor de marcadores de polimorfismo de un solo nucleótidos (SNP) que los disponibles actualmente por muchos servicios de genotipado.
Los investigadores de Aquaimpact demostraron mediante evaluaciones genómicas de supervivencia frente a la infección de la bacteria Flavobacterium columnare en la trucha arcoíris.
La creación de paneles de SNP que consistan en 3000-7000 SNP informativos sería una forma de reducir los costos de genotipado y adoptar la selección genómica en más peces reproductores en todo el mundo.
Selección genómica basada en miles de marcadores de ADN de todo el genoma
En los últimos años, un gran número de estudios han demostrado que la aplicación de la selección genómica mejora significativamente la respuesta a la selección de los programas de crianza selectiva acuícola.
En los programas de crianza selectiva en la acuicultura, la selección genómica se utiliza principalmente para las características que no se pueden medir directamente en los propios peces reproductores. Dichos rasgos son, por ejemplo, la calidad del producto, los rendimientos durante el procesamiento o los rasgos de resistencia a enfermedades que se miden en los descendientes (hermanos o medios hermanos) de los peces reproductores.
La selección genómica utiliza información de marcadores de ADN de todo el genoma, generalmente polimorfismo de un solo nucleótido (SNP), para estimar los valores genéticos genómicos de los candidatos de selección genotipados, en función de la información de genotipo y fenotipo recopila en sus hermanos y otros parientes (referidos como población de referencia).
Sin embargo, el costo de genotipado es un factor clave que limita el uso de la selección genómica en especies acuícolas. Para la trucha arcoíris, las evaluaciones genómicas se realizan comúnmente con un panel SNP que consta de alrededor de 57 000 marcadores SNP.
Resultados
Reducir el tamaño del panel de SNP a 7000 o incluso 3000 marcadores SNP en la trucha arcoíris solo redujo ligeramente la precisión en comparación con el panel completo de 28 000 marcadores SNP (Fig. 1).

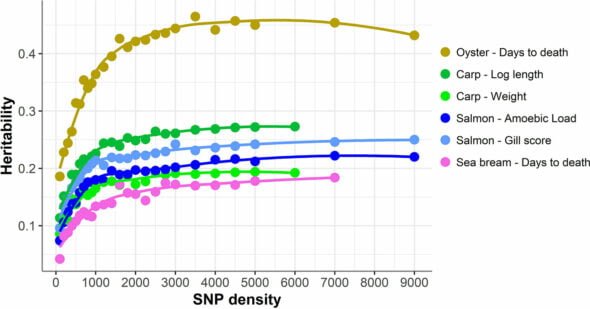
Se ha observado un resultado similar en una amplia variedad de especies acuícolas y para diferentes rasgos (Fig. 2). Esto significa que en la trucha arcoíris, el panel SNP comercial que se utiliza actualmente y que tiene 57 000 marcadores SNP es más que suficiente para una selección genómica precisa.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
El precio del genotipado depende de la cantidad de marcadores SNP analizados y de los volúmenes de individuos genotipados, cuantas más muestras para genotipar, menor será el precio.
Por lo tanto, una opción para reducir los costos de genotipado es generar un panel de marcadores SNP más pequeño, digamos 10 – 20 000. O tal vez desarrollar un panel de SNP multiespecies que pueda tener, por ejemplo, 20 000 marcadores SNP para cada una de las 3 especies diferentes (por ejemplo, 3 especies de salmónidos diferentes). Esto amplía considerablemente la base de clientes y reduce el precio del genotipado.
¿Por qué 3000 – 7000 SNP son suficientes para obtener la máxima precisión de selección en especies acuícolas?
El uso de 3000 – 7000 SNP para la selección genómica parece un número bajo, dado que en el ganado se utilizan cantidades mucho mayores de marcadores SNP. ¿Por qué esto sería así?
En los programas de cría selectiva en acuicultura, cada generación suele consistir en muchas familias grandes y de medio hermanos que se generan centralmente en el núcleo. Cada familia se divide y los hermanos de los futuros peces reproductores se analizan para detectar rasgos especiales como resistencia a enfermedades, calidad del producto o rasgos durante el sacrificio.
Los hermanos forman la población de referencia en la evaluación genómica y el vínculo entre su perfil de ADN y el rasgo por el que se miden se utiliza para estimar el valor de cría genómica del candidato de selección utilizando solo su perfil de ADN (los candidatos de selección no se miden por el rasgo).
Debido a que la población de referencia y los candidatos a reproducción están estrechamente relacionados, solo se necesita una cantidad moderada de marcadores SNP para capturar toda la variación genética y hacer una estimación precisa del valor de reproducción.
Además, en el programa de reproducción basado en la familia, dado que los peces están estrechamente relacionados, hay un número bajo de recombinación y, por lo tanto, lo más probable es que haya bloques de enlace largos en el genoma. Cuando el genoma consta de bloques de enlace largos, se necesitan menos marcadores SNP para cubrir todo el genoma y rastrear con precisión la relación entre los peces de la población de referencia y los candidatos.
En el ganado, en particular, la selección se práctica a través de múltiples generaciones y subpoblaciones, y los animales de la población de referencia tienen una relación más lejana con los candidatos a la reproducción. Por lo tanto, se espera que haya mucha recombinación en los conjuntos de datos y que el bloque de vinculación sea corto.
Socios involucrados
- The Roslin Institute and Royal (Dick) School of Veterinary Studies, University of Edinburgh, Edinburgh, United-Kingdom
- Natural Resources Institute Finland (Luke)
- Savon Taimen Oy
Referencias
Fraslin C, Koskinen H, Nousianen A, Houston RD, Kause A. 2022. Genome-wide association and genomic prediction of resistance to Flavobacterium columnare in a farmed rainbow trout population. Aquaculture 557: 738332. https://doi.org/10.1016/j.aquaculture.2022.738332
Fraslin C, Robledo D, Kause A, and Houston RD. 2023. Potential of imputation for cost-efficient genomic selection for resistance to Flavobacterium columnare in rainbow trout. https://doi.org/10.1101/2023.01.04.522693
Kriaridou C, Tsairidou S, Houston RD, Robledo D. Genomic prediction using low density marker panels in aquaculture: performance across species, traits, and genotyping platforms. Front Genet 2020;11. https://doi.org/10.3389/fgene.2020.00124
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.






