La industria de la acuicultura ha experimentado un crecimiento significativo en los últimos años, impulsado por la creciente demanda mundial de productos del mar. Sin embargo, el desarrollo de prácticas de acuicultura sostenibles y eficientes sigue siendo un desafío crítico.
La selección genómica (GS) y la selección asistida por marcadores (MAS) han surgido como herramientas poderosas para acelerar la mejora genética en las especies de acuicultura. Sin embargo, el costo de la genotipificación de SNP de alto rendimiento puede ser prohibitivo, en particular para especies con grandes tamaños de cría como el camarón.
Un equipo de científicos del Institute of Oceanology, de la University of Chinese Academy of Sciences, y del Qingdao Marine Science and Technology Center emplearon un método de secuenciación de análisis de segregación masiva (BSA-seq) para identificar polimorfismos de un solo nucleótido (SNP) y genes relacionados con el crecimiento en el camarón blanco del Pacífico Litopenaeus vannamei.
- 1 BSA-seq: una alternativa rentable
- 2 Aplicaciones de BSA-seq en la acuicultura
- 3 Identificación de SNP relacionados con el crecimiento en el camarón blanco del Pacífico
- 4 Análisis de datos y resultados
- 5 Análisis de enriquecimiento y genes candidatos
- 6 Implicaciones para la industria camaronera
- 7 Conclusión
- 8 Entradas relacionadas:
BSA-seq: una alternativa rentable
La secuenciación de análisis de segregación en masa (BSA-seq) ofrece una alternativa prometedora a los métodos tradicionales de genotipificación de SNP.
La BSA implica la combinación de muestras de ADN de individuos con fenotipos extremos (por ejemplo, tasas de crecimiento altas y bajas) e identificación de marcadores genéticos asociados con el rasgo de interés. Este método ofrece un ahorro de costos significativo en comparación con la genotipificación individual.
La combinación de BSA con la secuenciación de próxima generación (BSA-seq) ha revolucionado la identificación de marcadores relacionados con rasgos. Al analizar los datos de secuenciación de muestras de ADN combinadas, los investigadores pueden identificar variantes genéticas vinculadas a rasgos específicos.
Aplicaciones de BSA-seq en la acuicultura
BSA-seq se ha aplicado con éxito a varias especies de acuicultura, incluida L. vannamei. En este estudio, los investigadores utilizaron BSA-seq para identificar SNP y genes asociados con rasgos de crecimiento en L. vannamei. Al analizar dos familias independientes, pudieron minimizar el riesgo de falsos positivos e identificar marcadores genéticos con alta confianza.
Beneficios de BSA-seq para la cría en acuicultura
Los resultados del estudio publicado en la revista Aquaculture ofrecen varios beneficios para la cría en acuicultura:
- Entender la base genética de los rasgos de crecimiento: Los SNP y genes identificados brindan información valiosa sobre los mecanismos genéticos que subyacen al crecimiento del camarón.
- Selección asistida por marcadores (MAS): Los marcadores identificados se pueden utilizar en programas MAS para seleccionar individuos con características de crecimiento deseables.
- Selección genómica (GS): Los marcadores identificados también se pueden utilizar en programas GS para predecir los valores de cría de los individuos en función de su información genómica.
Identificación de SNP relacionados con el crecimiento en el camarón blanco del Pacífico
En el estudio, se empleó BSA-seq para identificar SNP y genes relacionados con el crecimiento en el camarón blanco del Pacífico Litopenaeus vannamei. Se utilizaron dos familias independientes para reducir la probabilidad de falsos positivos. Se agruparon y secuenciaron muestras de ADN de individuos con los pesos corporales más altos y más bajos.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Análisis de datos y resultados
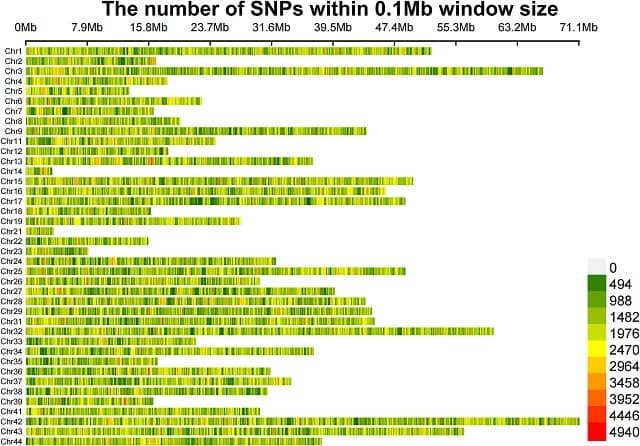
Se utilizaron dos métodos estadísticos, el método de la distancia euclidiana (ED) y la prueba de chi-cuadrado, para calcular las diferencias de alelos entre individuos con tasas de crecimiento altas y bajas. Se identificaron un total de 24.325.437 SNP y se encontró que 1.299 SNP y 163 genes estaban asociados con rasgos de crecimiento.
Análisis de enriquecimiento y genes candidatos
El análisis de enriquecimiento de KEGG reveló que los genes identificados estaban significativamente enriquecidos en las vías de señalización de fosfatidilinositol y fosfolipasa D. Varios genes involucrados en estas vías, incluyendo los genes de la diacilglicerol quinasa, los genes de la fosfatidilinositol 3-quinasa, los genes de la inositol polifosfato 5-fosfatasa, los genes de la proteína quinasa activada por mitógenos, los genes de la proteína relacionada con la miotubularina y los genes del receptor del factor de crecimiento epidérmico, fueron identificados como potenciales genes candidatos de crecimiento.
Implicaciones para la industria camaronera
Los resultados de este estudio proporcionan información valiosa sobre la base genética de los rasgos de crecimiento en el camarón blanco del Pacífico. Los SNP identificados se pueden utilizar para MAS y GS para acelerar la mejora genética en los programas de cría de camarones. Además, el estudio demuestra la eficacia y la rentabilidad de BSA-seq para la identificación de SNP en especies de acuicultura.
Conclusión
BSA-seq representa un enfoque prometedor para identificar SNP relacionados con el crecimiento en especies de acuicultura. Al aprovechar esta técnica, los investigadores pueden obtener una comprensión más profunda de la arquitectura genética de los rasgos importantes y desarrollar estrategias de cría más eficientes. Los hallazgos de este estudio contribuyen a los esfuerzos en curso para mejorar la sostenibilidad y la rentabilidad de la acuicultura del camarón.
El estudio fue financiado por Strategic Priority Research Program of the Chinese Academy of Sciences, National Key Research and Development Program of China, Key Research and Development Program of Shandong.
Contacto
Fuhua Li
Key Laboratory of Breeding Biotechnology and Sustainable Aquaculture(CAS), Institute of Oceanology, Chinese Academy of Sciences
Qingdao 266071, China.
Email: fhli@qdio.ac.cn
Referencia (acceso abierto)
Bao, Z., Yu, Y., Lin, P., & Li, F. (2025). Identification of growth-related genes based on BSA in Pacific white shrimp Litopenaeus vannamei. Aquaculture, 596, 741708. https://doi.org/10.1016/j.aquaculture.2024.741708
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.