El ADN ambiental (eDNA, por sus siglas en inglés) está revolucionando la forma en que los científicos estudian la biodiversidad, la conservación y la salud de los ecosistemas. Al analizar los rastros de ADN que se encuentran en el medio ambiente, los investigadores pueden monitorear las especies sin capturarlas ni perturbarlas directamente. Esta técnica está transformando la ecología, ofreciendo un método no invasivo y altamente eficiente para estudiar la vida silvestre, los ecosistemas acuáticos e incluso la biodiversidad antigua.
El ADN ambiental es la herramienta de biomonitoreo de más rápido crecimiento, impulsada por dos características clave: eficiencia temporal y sensibilidad (Takahashi et al., 2023). Pero, ¿Cuál es el potencial del ADN ambiental para la acuicultura?
La tecnología de ADNa ha surgido como una herramienta pionera en los campos de la pesca y la acuicultura, ofreciendo nuevos enfoques para el monitoreo y la gestión de los ecosistemas acuáticos (Chouhan et al., 2023). En este artículo, profundizaremos en qué es el ADN ambiental, cómo se recopila y analiza, sus aplicaciones en la investigación de la biodiversidad y su potencial para estimar la población de un estanques acuícola, o identificar potenciales patógenos.
- 1 ¿Qué es el ADN ambiental?
- 2 Cómo funciona el muestreo de ADN ambiental
- 3 ADN ambiental para la investigación y el monitoreo de la biodiversidad
- 4 Aplicación del ADN ambiental en la acuicultura
- 5 Ventajas y limitaciones del ADN ambiental
- 6 Futuro de la investigación y las aplicaciones del ADN ambiental
- 7 Conclusión
- 8 Referencias
- 9 Entradas relacionadas:
¿Qué es el ADN ambiental?
El ADN ambiental (eDNA) se refiere al material genético que los organismos liberan en su entorno, como el agua, el suelo, el aire o incluso el hielo. Este ADN proviene de varias fuentes, incluidas las células de la piel, la mucosidad, las heces y otros materiales biológicos. Al recopilar y analizar el eDNA, los científicos pueden detectar la presencia de especies sin capturarlas físicamente.
La tecnología ha avanzado rápidamente en los últimos años, lo que la convierte en una herramienta poderosa para la detección de especies, el monitoreo de ecosistemas y las evaluaciones de impacto ambiental. A diferencia de los métodos de muestreo tradicionales, que a menudo requieren la captura de individuos, el ADN ambiental proporciona un enfoque más ético y menos invasivo para estudiar la vida silvestre.
¿Por qué es importante el ADN ambiental?
- Monitoreo no invasivo: a diferencia de los estudios tradicionales que pueden requerir atrapar o etiquetar animales, el ADN ambiental permite a los científicos detectar especies sin interacción física. Takahashi et al., (2023) reportó que los métodos de ADNa hasta la fecha están «suficientemente avanzados para estudiar la composición de las comunidades de peces y reemplazar los métodos tradicionales más invasivos».
- Alta sensibilidad: incluso rastros diminutos de ADN pueden revelar la presencia de especies esquivas o raras.
- Amplias aplicaciones: el ADN ambiental se utiliza en la investigación de la biodiversidad, la biología de la conservación y la ecología forense. Al respecto, Ramey et al., (2024) reportó que el eDNA es una herramienta útil para detectar la presencia de salmón del Atlántico y del Pacífico y para comprender las amenazas a la salud de los peces y sus hábitats.
- Eficiencia en tiempo y costo: En comparación con los estudios de campo tradicionales, el análisis de ADN ambiental es más rápido y, a menudo, más barato.
- Estimación de biomasa: En la acuicultura el ADN ambiental se puede emplear para estimar el número de peces en un estanque acuícola.
Cómo funciona el muestreo de ADN ambiental
El muestreo de ADN ambiental implica recolectar material biológico de entornos naturales, como agua o suelo, y analizarlo en busca de marcadores genéticos. A continuación, se incluye una descripción general paso a paso del proceso:
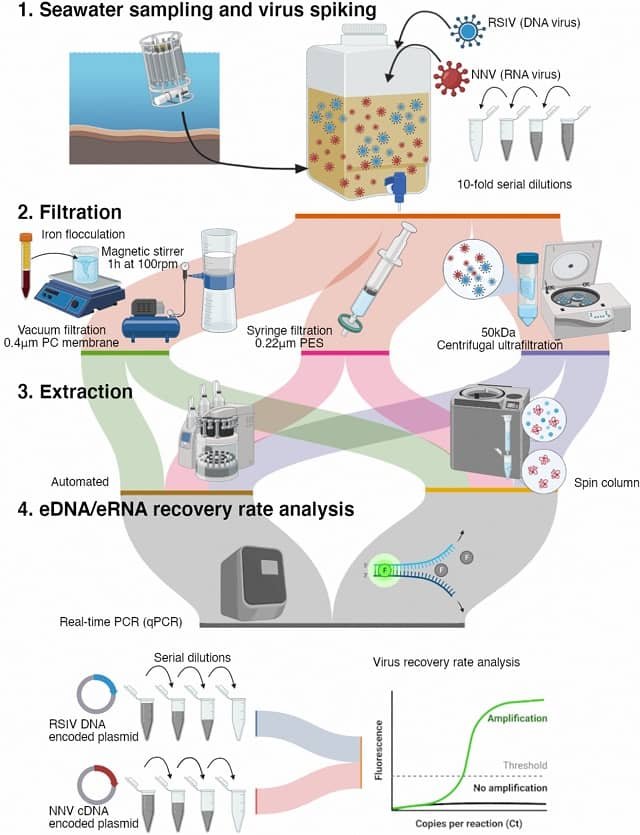
- Recolección de muestras: los científicos recolectan muestras ambientales (por ejemplo, agua, sedimento o aire) del campo utilizando técnicas estériles. Por ejemplo, Lee et al., (2024) destaca la eficacia de las redes de plancton como una herramienta simple y robusta basada en eDNA para la detección del VHSV (virus de la septicemia hemorrágica viral) en muestras de agua de acuicultura.
- Filtración y extracción de ADN: la muestra se filtra para concentrar el ADN, que luego se extrae utilizando protocolos de laboratorio.
- PCR (reacción en cadena de la polimerasa) o codificación de barras metabólica: se amplifican secuencias de ADN específicas para detectar especies objetivo.
- Secuenciación y análisis: el ADN se secuencia utilizando secuenciación de próxima generación (NGS) o PCR cuantitativa para identificar las especies presentes en la muestra.
- Interpretación de datos: las herramientas bioinformáticas comparan secuencias de ADN con bases de datos genéticas conocidas para determinar los organismos detectados.
ADN ambiental para la investigación y el monitoreo de la biodiversidad
El eDNA tiene el potencial de revolucionar la investigación y el monitoreo de la biodiversidad. Al respecto, Ge et al., (2025) introdujo el índice de integridad del ADN ambiental (eDI) para ajustar la concentración de eDNA con el propósito de estimar la biomasa de la carpa. El índice eDI se calcula como la relación entre el contenido de ADN de cadena larga y el contenido de ADN de cadena corta
Algunas de las aplicaciones clave del eDNA incluyen:
- Inventario de especies y mapeo de distribución: el eDNA puede usarse para identificar las especies presentes en un área en particular, incluso si son raras o esquivas.
- Detección temprana de especies invasoras: el eDNA puede usarse para detectar la presencia de especies invasoras en etapas tempranas, lo que permite una respuesta rápida y medidas de control.
- Monitoreo de tendencias poblacionales: el eDNA puede usarse para monitorear las tendencias poblacionales de las especies a lo largo del tiempo.
- Evaluación de la calidad del hábitat: el eDNA puede usarse para evaluar la calidad de los hábitats e identificar áreas que requieren esfuerzos de conservación.
- Detección de especies en peligro de extinción: el eDNA puede usarse para detectar especies en peligro de extinción, incluso en áreas remotas o inaccesibles.
Aplicación del ADN ambiental en la acuicultura
Estimación de la población
Momin et al., (2024) destaca que la concentración de eDNA podría ofrecer un método rápido y económico para medir la biomasa y/o abundancia, útil para evaluar poblaciones de peces. Al respecto, Benoit et al., (2023) encontró una relación positiva entre la concentración de ADN ambiental (eDNA) y la biomasa de salmón Chinook juvenil; sin embargo, el estudio también destaca que el uso de qPCR como método independiente para evaluar la biomasa de salmón juvenil podría ser solo semicuantitativo con los métodos actuales y requiere calibración adicional para su aplicación en el campo.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Monitoreo de la salud
De acuerdo con Momin et al., (2024) el monitoreo de eDNA probablemente tendrá un gran impacto en la capacidad de los productores de la industria salmonera y sus reguladores para detectar la presencia y abundancia de patógenos y otras amenazas biológicas en el entorno circundante. Krolicka et al., (2022) desarrolló un nuevo ensayo de qPCR específico para la detección de ADN ambiental (eDNA) del parásito del salmón Lepeophtheirus salmonis, que es compatible con el ESP (Environmental Sample Processor); este método representa una alternativa simple, no intrusiva y rentable para el monitoreo de L. salmonis en el campo y para proporcionar notificaciones rápidas de posibles infecciones a los productores sin causar problemas de bienestar a los peces.
Por su parte, Chouhan et al., (2023) y Ramey et al., (2024) informó que la aplicación de herramientas de eDNA también muestra ser prometedora para la detección temprana de patógenos emergentes o especies invasoras que podrían afectar negativamente la salud de los peces, con el potencial de promover una respuesta rápida y estrategias eficaces de mitigación y gestión.
Al respecto, Lo et al., (2023) presentó un enfoque de metabarcoding de ADN ambiental (eDNA), que emplea un modelo costero con una base de datos establecida, demostró un excelente potencial para el monitoreo diverso de patógenos costeros y reveló los mecanismos de prevalencia espacio-temporal asociados con la acuicultura intensa
Sin embargo, Aden et al., (2024) advierte que la implementación de herramientas de detección de ADNa/ARNa viral para la vigilancia de patógenos y la investigación de enfermedades en las granjas de peces de acuicultura requiere una cuidadosa consideración de la elección de los métodos de filtración de agua de mar.
Impacto ambiental
El ADN ambiental (ADNa) es una herramienta poderosa para detectar interacciones entre los ecosistemas de la acuicultura y las especies en ambientes bentónicos y pelágicos (English et al., 2024). En este sentido, Suzuki et al., (2024) utilizó el análisis de metabarcoding de ADN ambiental para determinar el impacto ambiental de la acuicultura del salmón Coho (Oncorhynchus kisutch) en la biodiversidad de peces en la bahía de Shizugawa, y reportó que el metabarcoding de ADN ambiental demostraron que la presencia o ausencia de la acuicultura explicó aproximadamente el 10% de la variación total en la composición de la comunidad de peces, una variación comparable a la de la profundidad del agua y el mes de muestreo.
Por su parte, Krkosek et al., (2024), mediante el uso del ADN ambiental, determinó que los patógenos provenientes de las granjas de salmón representan una preocupación para la conservación del salmón salvaje en Columbia Británica (BC). Específicamente, tres patógenos son prevalentes en el salmón Atlántico cultivado en BC y se transmiten al salmón del Pacífico salvaje, estando vinculados a impactos negativos en este último: Piscine orthoreovirus, Tenacibaculum spp. y piojos de mar (Lepeophtheirus salmonis).
Servicios ecosistémicos de las granjas acuícolas
Las granjas acuícolas de moluscos bivalvos proveen una serie de servicios ambientales. Mercaldo-Allen et al., (2021) encontró que la grabación de video y el metabarcoding de ADN ambiental (eDNA), aplicados en conjunto, proporcionaron un enfoque integral para describir los conjuntos de peces en la acuicultura de ostras estructurada y los hábitats de rocas que son difíciles de muestrear.
Por su parte, Li et al., (2024) empleó técnicas de ADN ambiental para identificar 55 especies de peces en la zona de cultivo de mejillones y en los arrecifes naturales circundantes de la isla Gouqi en el Mar de China Oriental, los investigadores reportaron que la diversidad en la zona de cultivo de mejillones fue significativamente mayor que en el hábitat de arrecifes naturales.
Ventajas y limitaciones del ADN ambiental
Ventajas del ADN ambiental
- No invasivo: reduce el estrés y los daños a la vida silvestre.
- Altamente sensible: detecta especies en densidades bajas.
- Escalable: se puede utilizar en diversos ecosistemas.
- Eficiente: ahorra tiempo y recursos en comparación con el muestreo tradicional.
Limitaciones del ADN ambiental
- Problemas de degradación: el ADN se degrada con el tiempo, lo que afecta la precisión.
- Riesgos de contaminación: requiere controles de laboratorio estrictos.
- Mareas y corrientes marinas pueden afectar la dispersión del ADNa.
- Presencia vs. Abundancia: detecta la presencia de especies, pero no siempre indica el tamaño de la población, o datos biométricos (tamaño, edad, sexo).
- Bases de datos incompletas: la identificación de especies depende de las bibliotecas de referencia genética existentes.
A pesar de estos desafíos, los avances en el protocolo de muestreo de ADN ambiental y la bioinformática están mejorando la confiabilidad de los estudios de ADN ambiental.
Futuro de la investigación y las aplicaciones del ADN ambiental
El futuro de la investigación del ADN ambiental parece prometedor, con innovaciones que amplían sus aplicaciones. Los científicos están desarrollando:
- Detección de ADN ambiental en tiempo real: dispositivos portátiles para análisis genético in situ. Deeg et al., (2025) desarrolló el sistema EZ-eDNA, el equipo está diseñado en torno a una caja de polímero protectora y utiliza componentes modulares que se pueden desplegar en diferentes arreglos utilizando diferentes configuraciones de bomba y filtro.
- ADN ambiental e inteligencia artificial: análisis impulsado por IA para una identificación de especies más rápida y precisa.
- Bibliotecas de referencia genómica: expansión de las bases de datos para una mejor correspondencia de especies.
A medida que avanza la tecnología, el ADN ambiental para la investigación y el monitoreo de la biodiversidad se volverá aún más preciso, accesible e impactante.
Conclusión
El ADN ambiental (eDNA) está revolucionando la ciencia medioambiental al ofrecer una forma poderosa, no invasiva y muy eficiente de estudiar la biodiversidad. Desde el seguimiento de especies en peligro de extinción hasta el control de la salud de los ecosistemas, el eDNA está dando forma al futuro de la conservación y la ecología.
A medida que la investigación continúa perfeccionando los protocolos de muestreo de ADN ambiental y ampliando sus aplicaciones, el eDNA seguirá estando a la vanguardia del control medioambiental y la conservación de especies. Al adoptar esta tecnología, los científicos, los responsables de las políticas y los conservacionistas pueden trabajar juntos para proteger la rica biodiversidad de nuestro planeta.
Referencias
Aden Ip, Y. C., Chen, J., Tan, L. Y., Lau, C., Chan, Y. H., Balasubramaniam, R. S., Jovinc Wong, W. Y., Ng, K., Brian Tan, Z. Y., Fernandez, C. J., Chang, S. F., & Yap, H. H. (2024). Establishing environmental DNA and RNA protocols for the simultaneous detection of fish viruses from seawater. Environmental DNA, 6(1), e418. https://doi.org/10.1002/edn3.418
Benoit, N. P., Robinson, K. M., E. Kellogg, C. T., Lemay, M. A., & V. Hunt, B. P. (2023). Using qPCR of environmental DNA (eDNA) to estimate the biomass of juvenile Pacific salmon (Oncorhynchus spp.). Environmental DNA, 5(4), 683-696. https://doi.org/10.1002/edn3.422
Chouhan, N., Dekari, D., Choudhary, B., Singh, A., & Choudhury, T. G. (2023). Environmental DNA (eDNA) technology: Fisheries and aquaculture perspectives. Indian J Anim Health, 62(2), 75-85.
Deeg, C. M., Saunders, R. G., Tam, C., Kaukinen, K., Li, S., Bass, A. L., & Miller, K. M. (2025). EDNA Sampling Systems for Salmon Ecosystem Monitoring. Environmental DNA, 7(1), e70059. https://doi.org/10.1002/edn3.70059
English, G., Lawrence, M. J., McKindsey, C. W., Lacoursière-Roussel, A., Bergeron, H., Gauthier, S., Wringe, B. F., & Trudel, M. (2024). A review of data collection methods used to monitor the associations of wild species with marine aquaculture sites. Reviews in Aquaculture, 16(3), 1160-1185. https://doi.org/10.1111/raq.12890
Ge, Q., Piao, Y., Li, Z., Yang, Y., Pan, M., & Bai, Y. (2025). Environmental DNA integrity index is sensitive for species biomass estimation in freshwater. Science of The Total Environment, 966, 178734. https://doi.org/10.1016/j.scitotenv.2025.178734
Krkosek, M., Bateman, A. W., Bass, A. L., Bugg, W. S., Connors, B. M., Deeg, C. M., Cicco, E. D., Godwin, S., Grimm, J., Krichel, L., Mordecai, G., Morton, A., Peacock, S., Shea, D., Riddell, B., & Miller, K. M. (2024). Pathogens from salmon aquaculture in relation to conservation of wild Pacific salmon in Canada. Science Advances. https://doi.org/adn7118
Krolicka A, Mæland Nilsen M, Klitgaard Hansen B, Wulf Jacobsen M, Provan F, Baussant T (2022) Sea lice (Lepeophtherius salmonis) detection and quantification around aquaculture installations using environmental DNA. PLoS ONE 17(9): e0274736. https://doi.org/10.1371/journal.pone.0274736
Lee, Y., Kang, H., Roh, H., Yun, D., Park, J., Lee, J., Heo, Y., Hong, S. Y., Jang, G., Kwon, M., Han, S., Kim, S., Park, C., & Kim, D. (2024). Enhancing viral detection in aquaculture water through virus-microplankton associations and plankton net concentration. Aquaculture, 590, 741092. https://doi.org/10.1016/j.aquaculture.2024.741092
Li, L., Zhang, S., & Wang, Z. (2024). EDNA technology reveals fish species diversity and ecological corridor function in large raft mussel aquaculture area in the East China Sea. Marine Pollution Bulletin, 209, 117171. https://doi.org/10.1016/j.marpolbul.2024.117171
Lo, L. S. H., Liu, X., Liu, H., Shao, M., Qian, P., & Cheng, J. (2023). Aquaculture bacterial pathogen database: Pathogen monitoring and screening in coastal waters using environmental DNA. Water Research X, 20, 100194. https://doi.org/10.1016/j.wroa.2023.100194
Mercaldo-Allen R, Clark P, Liu Y, Phillips G and others (2021) Exploring video and eDNA metabarcoding methods to assess oyster aquaculture cages as fish habitat. Aquacult Environ Interact 13:277-294. https://doi.org/10.3354/aei00408
Momin, C. M., Baidya, S., Debbarma, S., Yadav, N. K., Chandravashi, S., Debnath, A., … & Lavkush, V. A. (2024). Genetic improvement initiatives in aquaculture. Int J Adv Biochem Res, 8(4S), 441-447.
Ramey, A. M., McKeeman, C. M., Petrou, E. L., Menning, D. M., Russ, O. L., & López, J. A. (2024). Environmental DNA as a Tool for Better Understanding the Distribution, Abundance, And Health of Atlantic Salmon and Pacific Salmon. Fisheries, 49(4), 169-180. https://doi.org/10.1002/fsh.11038
Suzuki, S., Otomo, Y., Dazai, A., Abe, T., & Kondoh, M. (2024). Assessing the impacts of aquaculture on local fish communities using environmental DNA metabarcoding analysis. Environmental DNA, 6(3), e551. https://doi.org/10.1002/edn3.551
Takahashi, M., Saccò, M., Kestel, J. H., Nester, G., Campbell, M. A., Van der Heyde, M., Heydenrych, M. J., Juszkiewicz, D. J., Nevill, P., Dawkins, K. L., Bessey, C., Fernandes, K., Miller, H., Power, M., Mousavi-Derazmahalleh, M., Newton, J. P., White, N. E., Richards, Z. T., & Allentoft, M. E. (2023). Aquatic environmental DNA: A review of the macro-organismal biomonitoring revolution. Science of The Total Environment, 873, 162322. https://doi.org/10.1016/j.scitotenv.2023.162322
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.







