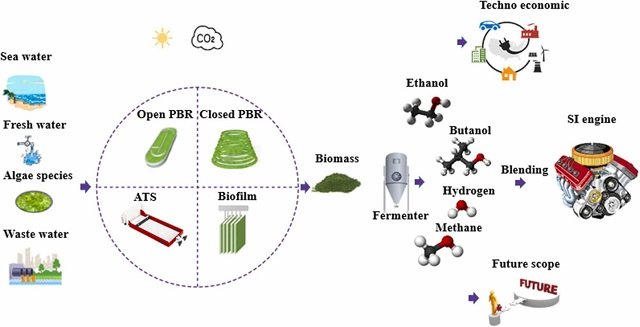
Brasil – Investigadores brasileños desarrollaron plataforma PhycoMine que almacena los datos moleculares de las microalgas Chlamydomonas reinhardtii y Chlorella vulgaris.
Las microalgas vienen recibiendo creciente atención de la comunidad científica debido a sus potenciales aplicaciones biotecnológicas y sus bioproductos.
Desde la liberación del secuenciamiento genómica de la especie modelo Chlamydomonas reihardtii, varias otros genomas de microalgas vienen siendo secuenciados y los datos genómicos funcionales contribuyen a expandir nuestro conocimiento sobre estos organismos y sus características evolutivas y metabólicas.
Recientemente se han desarrollado varias bases de datos para almacenar y minar los datos moleculares de las microalgas. Sin embargo, con el incremento de la disponibilidad de datos biológicos existe la necesidad por integrar las bases de datos.
En este sentido, los Investigadores de la Universidad de Sao Paulo desarrollaron PhycoMine, que es una base de datos con información biológica para integrar los datos de las microalgas de diferentes fuentes.
PhycoMine
PhycoMine es un sistema de almacenamiento de datos creado para acelerar el análisis de datos complejos e integrados de especies de microalgas en un solo ambiente computacional.
La plataforma ha sido desarrollado con el software InterMine, una herramienta de gestión del almacenamiento de base de datos biológicos, y tiene implementado una modelo de base de datos extendido, contiene una serie de herramientas que ayudan a los usuarios en el análisis y minería de datos individuales o grupos de datos.
Los investigadores procesaron los datos biológicos de diferentes fuentes para permitir la integración en la base de datos, incluido los datos del repositorio Phytozome para el registro de genes y transcritos, y data Uniprot para la información de proteínas anotadas.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Datos de Chlamydomonas reinhardtii y Chlorella vulgaris
En su primera versión la plataforma PhycoMine integra los datos disponibles de dos especies de microalgas: Chlamydomonas reinhardtii y Chlorella vulgaris.
Los principales tipos de datos integrados son genes anotados, proteínas, transcritos, gene, familias, ontología de genes, ortólogo de genes y datos de diferentes RNA-seq y experimentos de proteínas, entre otros tipos de información básica, tales como datos no publicados de modelos de respuestas fisiológicas clásicas como crecimiento celular, perfiles de consumo de nitrógeno y fosfato.
¿Cómo buscar la información?
Existen varias vías de acceso a la información almacenada en la base de datos, ya sea para obtener los datos de un solo elemento o de una lista de ellos.
Desde la pestaña de inicios, se encuentra disponible una herramienta de búsqueda específica para genes, proteínas y palabras clave, que permiten a los usuarios realizar una búsqueda directa usando identificadores de genes o proteínas, o varios de ellos separados por comas.
La búsqueda recupera una lista de elementos encontrados en la base de datos que coinciden con los identificadores de entrada. Asimismo, el usuario puede construir consultas personalizadas en la pestaña QueryBuilder.
Minería de datos y visualización
La minería de datos se puede realizar mediante listas. Se pueden insertar una lista de objetos (genes o proteínas) en la opción de “Analyze” en la página de inicio o se puede obtener de la base de datos PhycoMine cumpliendo ciertas restricciones.
PhycoMine cuenta con widgets creados para facilitar la minería de datos de forma simultánea de diferentes bases de datos.
Entre los widgets implementados en PhycoMine, existen opciones para la minería de la distribución de cromosomas, variación de la expresión de genes vía la transcriptómica, proteómica, Gene Onthology, KEGG, EggNOG, factores de transcripción y datos fenotípicos.
Los widgets fueron creados para facilitar la visualización de los datos de los niveles de expresión de los genes en diferentes configuraciones experimentales.
Finalmente el código de PhycoMine, así como el análisis y los scripts de alineamiento, y los datos de las microalgas Chlamydomonas reinhardtii y Chlorella vulgaris pueden ser encontrados en el repositorio labisapplication (https://github.com/labisapplication/Phycomine-features-Labis) en Github.
Otras plataformas para minar información sobre las microalgas incluyen a Ramanome, NanDeSyn para Nannochloropsis y dEMBF.
La plataforma PhycoMine es de libre acceso y puede ser visitada en: https://phycomine.iq.usp.br/
Contacto
Flavia Vischi Winck
winck@cena.usp.br,
Referencia (acceso libre):
Goitia R., D. Riaño, A. Fassio, F. Winck. 2021. PhycoMine: A Microalgae Data Warehouse. bioRxiv
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.