Una nueva investigación ha arrojado luz sobre el complejo rompecabezas genético de la resistencia a la Enfermedad del Páncreas (PD) en el salmón del Atlántico. Científicos han identificado una región específica en el genoma del salmón y tres genes candidatos que son decisivos en la respuesta del pez al alfavirus salmónido (SAV), el agente causante de esta devastadora enfermedad.
Estos hallazgos, publicados en la revista BMC Genomics, por científicos de la Norwegian University of Life Sciences, de AquaGen AS y de la University of Aberdeen, no solo confirman la importancia de la genética en la lucha contra la PD, sino que también abren la puerta al desarrollo de estrategias de manejo y programas de selección genómica mucho más precisos y eficaces para la industria salmonicultora.
- 1 Un desafío persistente para la salmonicultura
- 2 Mapeando la resistencia: una inmersión genómica profunda
- 3 Identificando los genes de la resistencia a PD
- 4 Una paradoja inesperada: más expresión génica, menor resistencia
- 5 Implicaciones para la acuicultura: hacia una selección genómica más precisa
- 6 Conclusión: un futuro más resiliente para el salmón
- 7 Entradas relacionadas:
Un desafío persistente para la salmonicultura
La Enfermedad del Páncreas es uno de los mayores desafíos sanitarios para el cultivo de salmón del Atlántico. Causada por el alfavirus salmónido (SAV), esta patología provoca una inflamación severa y necrosis del páncreas, lo que lleva a la pérdida de apetito, letargo, y una mayor susceptibilidad a infecciones secundarias.
Los brotes de PD pueden generar tasas de mortalidad de hasta un 28% y, aunque algunos peces logran sobrevivir, a menudo quedan con un crecimiento retrasado, lo que impacta negativamente en la producción y el bienestar animal. Si bien las vacunas y la cría selectiva han ayudado a mitigar el problema, su eficacia es solo parcial. Por ello, comprender a fondo la base genética de la resistencia es fundamental para desarrollar soluciones sostenibles.
Mapeando la resistencia: una inmersión genómica profunda
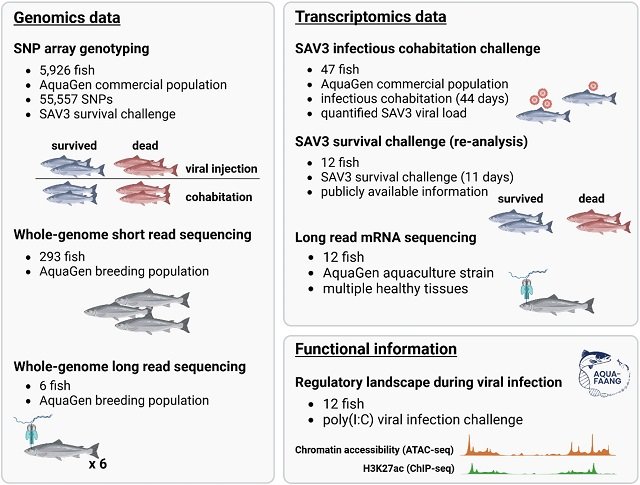
Para descifrar el código genético de la resistencia a la PD, los investigadores llevaron a cabo un estudio a gran escala, utilizando tecnologías genómicas de vanguardia.
El estudio analizó datos de genotipo y supervivencia de 5,628 salmones del Atlántico provenientes de tres ensayos de desafío viral independientes realizados entre 2018 y 2020. Para simular condiciones diversas, los peces fueron infectados con el subtipo SAV3, el más agresivo en Noruega, mediante dos métodos distintos:
- Inyección peritoneal (IP): Un método de infección directa y controlado.
- Cohabitación (CH): Un método que imita la transmisión natural del virus en un centro de cultivo, donde peces sanos comparten agua con individuos infectados.
Potenciando el análisis con genómica avanzada
El principal avance de este estudio fue el uso de técnicas de imputación de genotipos. Los investigadores partieron de un panel de aproximadamente 55,000 marcadores genéticos (SNPs) y, utilizando un conjunto de referencia de 293 genomas completos, lograron inferir y expandir el conjunto de datos a más de 938,000 SNPs. Este «mapa» genético de alta resolución permitió una localización mucho más precisa de las regiones asociadas con la resistencia a la enfermedad.
Además, se utilizaron tecnologías de secuenciación de lectura larga (long-read) para identificar variantes genéticas más complejas, como inserciones, deleciones y microsatélites, que las tecnologías estándar a menudo no logran detectar.
Identificando los genes de la resistencia a PD
El análisis de esta enorme cantidad de datos genómicos condujo a descubrimientos reveladores sobre la arquitectura genética de la respuesta a la PD.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Dos regiones genéticas (QTL) en el foco, pero una es la protagonista
El estudio confirmó la presencia de dos Loci de Rasgos Cuantitativos (QTL), que son regiones del genoma que afectan a un rasgo particular, asociados con la resistencia a la PD:
- Un QTL principal en el cromosoma Ssa03: Esta región mostró una asociación muy fuerte con la supervivencia a la enfermedad, independientemente del método de infección (IP o CH).
- Un QTL secundario en el cromosoma Ssa07: Curiosamente, esta región solo mostró una asociación significativa en los peces infectados por inyección directa (IP), pero no en los de cohabitación. Esto sugiere que el QTL de Ssa07 podría estar involucrado en una respuesta inmune específica que se activa por la ruta de infección artificial, mientras que el QTL de Ssa03 es el actor principal en la resistencia general a la enfermedad.
Tres genes «gig1-like»: los candidatos principales
Gracias a la alta resolución del análisis, los investigadores lograron reducir la vasta región del QTL principal en Ssa03 a un fragmento de solo 45 kilobases (kb). Dentro de esta pequeña porción del genoma se encuentran seis genes.
Para determinar cuál de estos era el verdadero responsable, se analizaron datos de expresión génica (transcriptómica) de peces con altas y bajas cargas virales. Los resultados fueron contundentes: solo tres copias de un gen conocido como gig1-like mostraron una respuesta significativa a la infección por SAV3. Esto los posiciona como los candidatos causales más probables para la resistencia a la PD.
Una paradoja inesperada: más expresión génica, menor resistencia
Uno de los hallazgos más intrigantes del estudio es la relación entre la expresión de estos genes y la enfermedad. Contrario a lo que se podría esperar, los análisis mostraron que una mayor expresión de los genes gig1-like se correlacionaba con una mayor carga viral y una menor supervivencia de los peces.
Esto sugiere que los genes gig1-like, que son inducidos por la respuesta inmune del pez al virus, podrían funcionar más como un indicador de la replicación viral que como un mecanismo de supresión viral. En otras palabras, una alta actividad de estos genes parece ser una señal de que el virus está ganando la batalla en el organismo del pez.
Implicaciones para la acuicultura: hacia una selección genómica más precisa
Estos descubrimientos tienen un potencial transformador para los programas de mejora genética en la acuicultura del salmón.
La identificación de los tres genes gig1-like y de variantes genéticas específicas dentro de ellos —incluyendo polimorfismos de un solo nucleótido (SNPs) y microsatélites— proporciona a los genetistas objetivos mucho más específicos y fiables para la selección. En lugar de seleccionar basándose en una región QTL amplia y poco definida, ahora es posible centrarse en los marcadores genéticos que tienen un impacto funcional directo en la respuesta a la enfermedad.
La incorporación de esta información en los programas de cría permitirá seleccionar reproductores con las variantes genéticas asociadas a una mayor resistencia, acelerando el progreso genético y contribuyendo a la producción de poblaciones de salmón más robustas y saludables.
Conclusión: un futuro más resiliente para el salmón
Este estudio representa un paso de gigante en nuestra comprensión de la base genética de la resistencia a la Enfermedad del Páncreas. Al combinar datos de desafíos virales a gran escala con las herramientas genómicas más avanzadas, los investigadores han pasado de identificar amplias regiones genómicas a señalar genes específicos y las variantes dentro de ellos que orquestan la respuesta a la infección por SAV.
El camino hacia salmones completamente resistentes a la PD aún es largo, pero estos hallazgos proporcionan un mapa detallado y preciso que, sin duda, fortalecerá las estrategias de manejo de enfermedades y promoverá una acuicultura más sostenible y resiliente.
Contacto
Domniki Manousi
Centre for Integrative Genetics, Department of Animal and Aquacultural Sciences, Faculty of Biosciences, Norwegian University of Life Sciences
Oluf Thesens vei 6, Ås, 1433, Norway
Email: domniki.manousi@nmbu.no
Referencia (acceso abierto)
Manousi, D., Jaskula, D.M., Grammes, F. et al. Dissecting the genetic basis of response to salmonid alphavirus in Atlantic salmon. BMC Genomics 26, 657 (2025). https://doi.org/10.1186/s12864-025-11735-2
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.







