Los sistemas de recirculación acuícola (RAS) son pilares de la acuicultura sostenible, pero la alta densidad de cultivo los hace vulnerables a brotes de enfermedades que pueden propagarse rápidamente. Identificar al agente causante a tiempo es crucial, pero los métodos tradicionales, como los cultivos y el PCR, a menudo «enmascaran» la verdadera diversidad bacteriana y la prevalencia de patógenos.
Un estudio reciente, publicado por investigadores del Interdisciplinary Center for Aquaculture Research (INCAR) de la University of Concepción, de la Universidad San Sebastián y de MOWI Chile SA, motivado por un evento de mortalidad en un RAS comercial de salmón del Atlántico (Salmo salar) en Chile, ha empleado la tecnología de secuenciación Oxford Nanopore (ONT) para obtener un perfil microbiano de alta definición y descubrir al culpable.
Los hallazgos revelan no solo al patógeno responsable, sino también las diferencias funcionales en la microbiota intestinal que separan a un pez sano de uno enfermo.
- 1 Conclusiones clave
- 2 El desafío: un brote de origen desconocido
- 3 Una nueva herramienta: secuenciación 16s de gen completo
- 4 Hallazgos clave: la microbiota del salmón enfermo vs. el sano
- 5 Más allá de quién está: qué «hacen» las bacterias
- 6 El rol del sistema: los biofiltros como reservorios
- 7 Conclusión: una herramienta proactiva para la salud del ras
- 8 Entradas relacionadas:
Conclusiones clave
- Los salmones del Atlántico que presentaban hinchazón abdominal (enfermos) mostraron una diversidad microbiana intestinal significativamente menor en comparación con los peces sin síntomas clínicos.
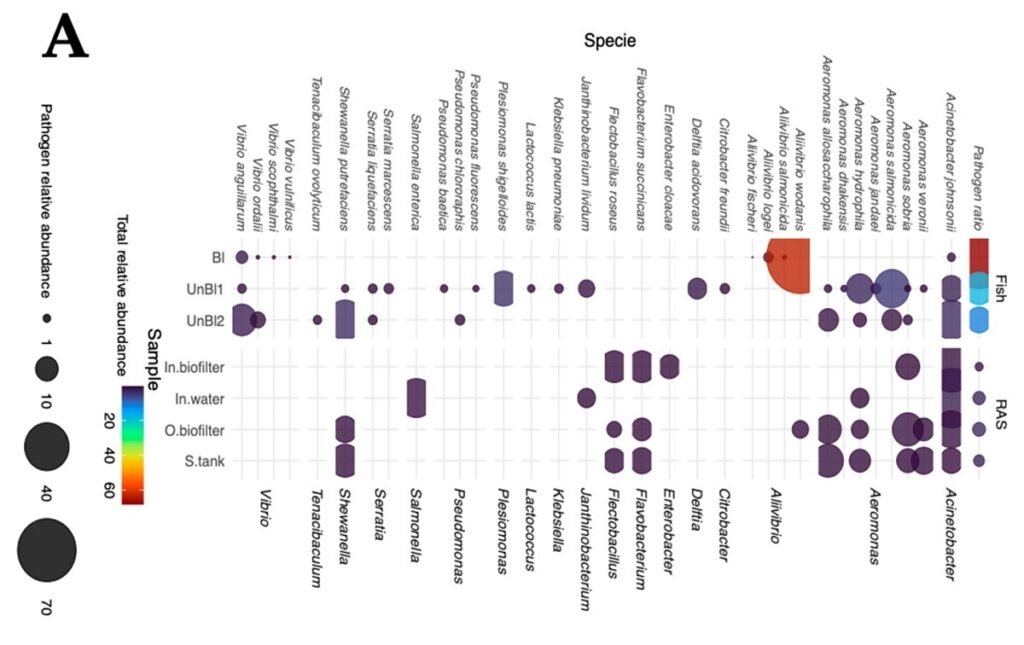
- Se detectó una alta abundancia del patógeno Aliivibrio wodanis en los intestinos de los salmones enfermos. Notablemente, esta bacteria estaba ausente en los peces sanos.
- Los peces sanos mostraron más vías metabólicas ligadas a la producción de aminoácidos y ácidos grasos de cadena corta, cruciales para la buena salud. Los peces enfermos, en cambio, mostraron un aumento en el metabolismo del azufre (producción de H2S), una función asociada directamente a la presencia de A. wodanis.
- Se detectó A. wodanis en la salida del biofiltro del RAS, lo que sugiere que los biofilms del sistema pueden actuar como reservorios para este patógeno, permitiendo la contaminación cruzada entre el agua y los peces.
- La secuenciación Nanopore del gen 16S completo ofrece una mayor resolución taxonómica (identificación a nivel de especie) y resultados más rápidos (en ~24h) que los métodos tradicionales de PCR o secuenciación de lecturas cortas, siendo ideal para la vigilancia sanitaria.
El desafío: un brote de origen desconocido
El estudio se inició tras un evento de mortalidad en una piscicultura comercial. Los salmones afectados mostraban signos clínicos claros: distensión abdominal (hinchazón) y diversos grados de peritonitis e inflamación granulomatosa.
El personal de la instalación sospechó inicialmente de patógenos conocidos como Rhodococcus sp. o Francisella sp., pero las pruebas de PCR para estos agentes resultaron negativas. Este callejón sin salida diagnóstico subrayó la necesidad de un enfoque más profundo y amplio que pudiera analizar a toda la comunidad bacteriana presente.
Una nueva herramienta: secuenciación 16s de gen completo
Los investigadores recurrieron a la metagenómica basada en Nanopore. A diferencia de las técnicas de secuenciación más comunes que solo leen pequeñas regiones variables del gen 16S rRNA (como V3-V4), la tecnología Nanopore permite secuenciar el gen 16S completo (V1-V9).
Esta capacidad de «lectura larga» ofrece una resolución taxonómica mucho mayor, permitiendo identificar bacterias a nivel de especie de forma más fiable.
“Una de las ventajas de Nanopore es que permite leer el gen 16S completo, identificando bacterias hasta nivel de especie, algo que no se logra con otras tecnologías de secuenciación. Además, es rápida y portátil: en menos de un día podemos tener un panorama completo de la comunidad microbiana, incluyendo patógenos que los métodos tradicionales podrían pasar por alto”, explicó el Investigador Adjunto de la línea “Genómica Acuícola” de INCAR, Dr. Diego Valenzuela-Miranda.
Para el estudio, los investigadores tomaron muestras de intestino de peces enfermos (hinchados) y peces sanos (sin síntomas), así como muestras de agua de componentes clave del RAS: la entrada de agua, el tanque de sedimentación y los biofiltros.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Hallazgos clave: la microbiota del salmón enfermo vs. el sano
Los principales resultados de los investigadores revelaron la existencia de bacterias relacionadas con procesos de desnitrificación y metabolismo del sulfuro de hidrógeno en el agua. Ellos determinaron que los peces enfermos mostraron menor diversidad microbiana y predominancia de bacterias del grupo Proteobacteria. Además, se detectaron patógenos como Aeromonas, Vibrio y Aliivibrio, incluyendo Aliivibrio wodanis en peces con síntomas clínicos. En contraste, los salmones saludables presentaron rutas metabólicas asociadas a una mejor digestión y fermentación de nutrientes.
El análisis de secuenciación arrojó luz inmediata sobre la condición de los peces. El hallazgo más evidente fue que los peces enfermos presentaban una diversidad microbiana intestinal drásticamente reducida en comparación con los peces sanos, un conocido indicador de disbiosis y mala salud.
El dominio de proteobacteria y un patógeno clave
Al analizar la composición, la diferencia fue abrumadora. Los intestinos de los peces enfermos estaban dominados en más de un 98% por el filo Proteobacteria.
Dentro de este grupo, un patógeno emergió como el principal sospechoso: Aliivibrio wodanis. Esta bacteria se encontró en altas concentraciones en los peces con distensión abdominal, pero lo más revelador es que estaba completamente ausente en las muestras de peces sanos.
Curiosamente, A. wodanis se asocia clásicamente con la «enfermedad de la úlcera de invierno», pero los síntomas observados en este brote (hinchazón y peritonitis) eran diferentes. Esto sugiere que la patogenicidad de A. wodanis puede variar o manifestarse de forma distinta según las condiciones del sistema.
Patógenos oportunistas: un equilibrio delicado
El estudio también demostró que la salud no es simplemente la ausencia de patógenos. Bacterias de géneros potencialmente patógenos como Aeromonas y Vibrio (incluyendo V. anguillarum) se detectaron en todos los peces, tanto sanos como enfermos.
Esto refuerza la idea de que muchos patógenos son oportunistas. En un intestino sano y diverso, su presencia está controlada. Sin embargo, en un estado de disbiosis (baja diversidad), estos oportunistas, o patógenos emergentes como A. wodanis, pueden proliferar y causar la enfermedad.
Más allá de quién está: qué «hacen» las bacterias
La metagenómica no solo identifica quién está presente, sino que también puede predecir qué funciones metabólicas está realizando esa comunidad. Aquí, las diferencias funcionales fueron tan marcadas como las taxonómicas.
El perfil funcional de un pez sano
La microbiota de los peces sanos mostró una mayor contribución de vías metabólicas relacionadas con:
- Metabolismo de aminoácidos.
- Fermentación de ácidos grasos de cadena corta (AGCC).
Ambas vías son fundamentales para la nutrición, el metabolismo energético y el bienestar general del pez.
El perfil funcional de un pez enfermo
Por el contrario, la microbiota de los peces enfermos mostró un aumento significativo en las vías relacionadas con el metabolismo del azufre.
Específicamente, esto se tradujo en una mayor producción de sulfuro de hidrógeno (H2S). El análisis funcional identificó a A. wodanis como el principal contribuyente a esta producción de H2S en los peces enfermos.
El exceso de H2S en el intestino es problemático; no solo es un compuesto tóxico, sino que también puede tener efectos citoprotectores para las propias bacterias, ayudándolas a resistir los antibióticos y complicando el tratamiento.
El rol del sistema: los biofiltros como reservorios
El estudio no se limitó a los peces. El análisis del agua del RAS reveló que A. wodanis también se detectó en la salida del biofiltro.
“Detectamos Aliivibrio wodanis en peces enfermos y también en el agua del sistema, lo que sugiere una posible transmisión ambiental. Esto demuestra que los biofiltros y tanques pueden actuar como reservorios de patógenos, por lo que es clave monitorear no solo los peces, sino también el entorno microbiológico del RAS”, detalló el Dr. Valenzuela-Miranda.
Este es un hallazgo crítico para la gestión de los RAS. Demuestra que los biofilms que crecen en los componentes del sistema (como los biofiltros) pueden actuar como reservorios de patógenos. Desde allí, las bacterias pueden desprenderse, permanecer en la columna de agua y causar la recolonización o infección de los peces, creando un ciclo de contaminación cruzada.
Conclusión: una herramienta proactiva para la salud del ras
Este estudio demuestra el poder de la secuenciación Nanopore de gen 16S completo como una herramienta de diagnóstico y vigilancia rápida y precisa en la acuicultura.
- Logró donde el PCR tradicional falló: identificó a Aliivibrio wodanis como el patógeno putativamente responsable del brote, no solo por su presencia en peces enfermos, sino también por su función metabólica perjudicial (producción de H2S).
Estos hallazgos sugieren que el uso de tecnologías como Nanopore puede ayudar a monitorear de forma más precisa la salud microbiana en sistemas de cultivo, permitiendo detectar problemas antes de que afecten la producción.
“Si incorporamos esta tecnología al monitoreo rutinario, podríamos anticipar brotes y actuar preventivamente, reduciendo el uso de antibióticos y mejorando la salud de los peces y del sistema”, finalizó el investigador.
Contacto
Diego Valenzuela-Miranda
Interdisciplinary Center for Aquaculture Research (INCAR), University of Concepción
Concepción 4070409, Chile
Email: divalenzuela@udec.cl
Referencia (acceso abierto)
Valenzuela-Miranda, D., Morales-Rivera, M., Mancilla-Schutz, J., Sandoval, A., Valenzuela-Muñoz, V., & Gallardo-Escárate, C. (2025). Nanopore-Based Metagenomic Approaches for Detection of Bacterial Pathogens in Recirculating Aquaculture Systems. Fishes, 10(10), 496. https://doi.org/10.3390/fishes10100496
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.







