Corvalis, EEUU.- Los científicos de la US Geological Survey (USGS) y de la Washington State University (WSU) han descubierto que el salmón Chinook puede ser detectado por el ADN que ellos liberan en el ambiente. Los resultados son parte de un tema especial de la revista Biological Conservation sobre el uso del ADN ambiental (eADN) para la conservación y la gestión de las especies acuáticas.
El número especial de la revista contiene doce informes científicos sobre la detección de especies acuáticas usando el eADN, desde el concepto hasta la práctica, e incluye una evaluación de los beneficios potenciales, limitaciones y sesgos de aplicar los métodos de eADN para investigar y monitorear los animales.
“Los informes en esta edición especial demuestran que las técnicas de eADN están empezando a hacer una real contribución al campo de la biología de la conservación en todo el mundo” dijo Caren Goldberg, profesor en la Washington State University y editora del número especial de la revista.
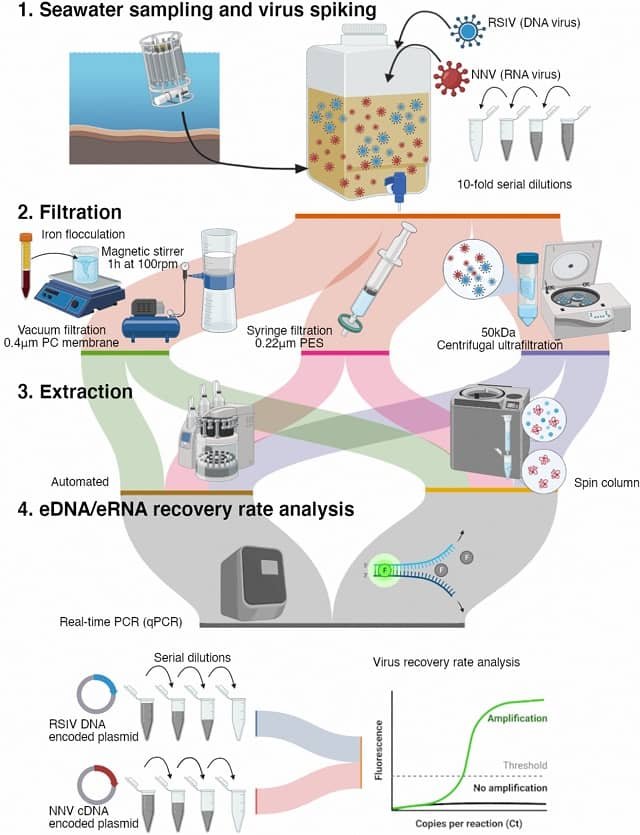
El ADN, el ácido desoxirribonucleico, es el material hereditario que contiene las instrucciones biológicas para construir y mantener todas las formas de vida; el eADN es el ADN que los animales liberan en el ambiente a través de procesos biológicos normales de fuentes como heces, mucus, piel, pelos y cadáveres. La investigación y el monitoreo de especies raras, en peligro de extinción e invasoras puede ser realizado mediante el análisis del eADN en las muestras de agua.
Un informe incluye los resultados de la investigación de los ecologistas de USGS, Matthew Laramie y David Pilliod, en donde evalúan el potencial del análisis de eADN para mejorar la detección del salmón Chinook en el río Columbia. Esta es la primera vez que los métodos eADN son usados para monitorear las poblaciones de salmón de América del Norte.
“Los resultados de este estudio indican que los métodos de detección de eADN son una vía efectiva para determinar la distribución del Chinook a través de una gran área y pueden ser potencialmente usados para documentar el arribo de las especies migratorias, como el salmón del Pacífico, o la colonización de los arroyos después de la restauración del hábitat o los esfuerzos de reintroducción” dijo Laramie.
Los informes de la edición especial de la revista se concentran en las técnicas para analizar las muestras de eADN, la producción y degradación de eADN en el ambiente y el laboratorio, y las aplicaciones prácticas de las técnicas de eADN en la detección y gestión de los peces y anfibios en peligro de extinción.
Katherine Strickler, científica de la WSU, apertura la edición especial con una revisión del estado de la ciencia del eADN, un campo desarrollado en base a estudios de microorganismos en las muestras del ambiente y el ADN recolectado de especímenes antiguos como tejidos momificados o plantas preservadas.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Por otro lado, el Profesor Alexander Fremier de la WSU, y otros científicos, cuantificaron los efectos de la radicación ultravioleta, temperatura y pH en la degradación del eADN en los sistemas acuático.
“Necesitamos conocer mejor por cuanto tiempo el ADN puede ser detectado en el agua bajo diferentes condiciones. Nuestro trabajo ayudará a mejorar las estrategias de muestreo para el monitoreo del eADN de especies sensible e invasoras” dijo Strickler.
Contacto:
U.S. Department of the Interior, U.S. Geological Survey
Office of Communications and Publishing
12201 Sunrise Valley Dr, MS 119
Reston, VA 20192
Susan Kemp
skemp@usgs.gov
Phone: 541-750-1047
Paul Laustsen
plaustsen@usgs.gov
Phone: 650-329-4046
Referencia:
Takahara T., T. Minamoto and H. Doi. 2014. Effects of sample processing on the detection rate of environmental DNA from the Common Carp (Cyprinus carpio). Biological Conservation. doi:10.1016/j.biocon.2014.11.014.
http://www.sciencedirect.com/science/article/pii/S000632071400439X
Deiner K., J. Walser, E. Mächler and F. Altermatt. 2014. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA. Biological Conservation. doi:10.1016/j.biocon.2014.11.018
http://www.sciencedirect.com/science/article/pii/S0006320714004431
Stephen F. Spear, John D. Groves, Lori A. Williams, Lisette P. Waits. 2014. Using environmental DNA methods to improve detectability in a hellbender (Cryptobranchus alleganiensis) monitoring program. Biological Conservation. doi:10.1016/j.biocon.2014.11.016
http://www.sciencedirect.com/science/article/pii/S0006320714004418
Cameron R. Turner, Karen L. Uy, Robert C. Everhart. 2014. Fish environmental DNA is more concentrated in aquatic sediments than surface water. Biological Conservation. doi:10.1016/j.biocon.2014.11.017 (Acceso abierto)
http://www.sciencedirect.com/science/article/pii/S000632071400442X
Katy E. Klymus, Catherine A. Richter, Duane C. Chapman, Craig Paukert. 2014. Quantification of eDNA shedding rates from invasive bighead carp Hypophthalmichthys nobilis and silver carp Hypophthalmichthys molitrix. Biological Conservation. doi:10.1016/j.biocon.2014.11.020
http://www.sciencedirect.com/science/article/pii/S0006320714004455
Anna M. McKee, Stephen F. Spear, Todd W. Pierson. 2014. The effect of dilution and the use of a post-extraction nucleic acid purification column on the accuracy, precision, and inhibition of environmental DNA samples. Biological Conservation. doi:10.1016/j.biocon.2014.11.031
http://www.sciencedirect.com/science/article/pii/S000632071400456X
Matthew B. Laramie, David S. Pilliod, Caren S. Goldberg. 2014. Characterizing the distribution of an endangered salmonid using environmental DNA analysis. Biological Conservation. doi:10.1016/j.biocon.2014.11.025 (Acceso abierto)
http://www.sciencedirect.com/science/article/pii/S0006320714004509
Caren S. Goldberg, Katherine M. Strickler, David S. Pilliod. 2014. Moving environmental DNA methods from concept to practice for monitoring aquatic macroorganisms. Biological Conservation. doi:10.1016/j.biocon.2014.11.040 (Acceso abierto)
http://www.sciencedirect.com/science/article/pii/S0006320714004650
Eva Egelyng Sigsgaard, Henrik Carl, Peter Rask Møller, Philip Francis Thomsen. 2014. Monitoring the near-extinct European weather loach in Denmark based on environmental DNA from water samples. Biological Conservation. doi:10.1016/j.biocon.2014.11.023
http://www.sciencedirect.com/science/article/pii/S0006320714004480
Jeremy Biggs, Naomi Ewald, Alice Valentini, Coline Gaboriaud, Tony Dejean, Richard A. Griffiths, Jim Foster, John W. Wilkinson, Andy Arnell, Peter Brotherton, Penny Williams, Francesca Dunn. 2014. Using eDNA to develop a national citizen science-based monitoring programme for the great crested newt (Triturus cristatus). Biological Conservation. doi:10.1016/j.biocon.2014.11.029
http://www.sciencedirect.com/science/article/pii/S0006320714004546
Philip Francis Thomsen, Eske Willerslev. 2014. Environmental DNA – An emerging tool in conservation for monitoring past and present biodiversity. Biological Conservation. doi:10.1016/j.biocon.2014.11.019.
http://www.sciencedirect.com/science/article/pii/S0006320714004443
Katherine M. Strickler, Alexander K. Fremier, Caren S. Goldberg. 2014. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms. Biological Conservation. doi:10.1016/j.biocon.2014.11.038
http://www.sciencedirect.com/science/article/pii/S0006320714004637
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.