El éxito en la producción a gran escala de microalgas, como Desmodesmus armatus, no solo depende de la luz, los nutrientes o el diseño del reactor. Existe un universo microscópico invisible, un ecosistema complejo conocido como microbioma, que vive junto a las algas y juega un papel decisivo en la salud y productividad del cultivo. Un estudio reciente, publicado en Bioresource Technology por científicos de Institute of Marine Sciences (ICM), de la Universidad de Murcia y de la Universidad de Almería, ha profundizado en esta intrincada red de interacciones para desvelar qué microorganismos actúan como aliados y cuáles como enemigos.
Los hallazgos ofrecen una valiosa hoja de ruta para optimizar los procesos de producción industrial, especialmente en áreas tan prometedoras como el tratamiento de aguas residuales y la generación sostenible de biomasa.
- 1 El universo microbiano en los reactores raceways
- 2 ¿Quién vive junto a Desmodesmus armatus?
- 3 Identificando a los «buenos»: bacterias que promueven un cultivo saludable
- 4 Detectando a los «malos»: patógenos bacterianos y fúngicos
- 5 Implicaciones para el futuro de la producción de microalgas
- 6 Entradas relacionadas:
El universo microbiano en los reactores raceways
Las microalgas son una piedra angular de la bioeconomía circular, capaces de convertir luz solar y CO2 en bioplásticos, biocombustibles y suplementos nutricionales de alto valor. Sin embargo, llevar su producción a escala industrial enfrenta un desafío crítico: la estabilidad del cultivo. Las caídas de productividad o el colapso total de un cultivo a menudo se deben a desequilibrios en su microbioma, donde microorganismos dañinos ganan terreno sobre los beneficiosos.
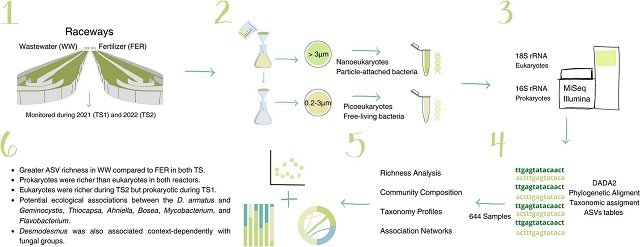
Para entender esta dinámica, un equipo de investigación monitoreó durante dos períodos de 8 meses (en 2021 y 2022) las comunidades microbianas en dos reactores de tipo raceway a gran escala, ambos inoculados con la microalga Desmodesmus armatus. La principal diferencia entre ellos era su «dieta»: uno se alimentaba con aguas residuales urbanas (WW), mientras que el otro utilizaba agua limpia suplementada con fertilizantes (FER).
Mediante la técnica de metabarcoding de ARNr 16S y 18S, los científicos analizaron la diversidad y composición de las comunidades de procariotas (bacterias) y eucariotas (como hongos y otros protistas) que crecían junto a la microalga.
¿Quién vive junto a Desmodesmus armatus?
El estudio reveló una diversidad microbiana asombrosa y compleja. Algunos de los hallazgos más destacados sobre la composición de estas comunidades fueron:
- Mayor riqueza en aguas residuales: El reactor que utilizaba aguas residuales (WW) mostró una riqueza de especies (número de variantes genéticas o ASVs) significativamente mayor que el reactor con fertilizantes (FER), tanto para bacterias como para eucariotas. Esto sugiere que la composición del agua de entrada es un factor clave que moldea el microbioma.
- Dominancia bacteriana: En ambos reactores, la riqueza de bacterias fue consistentemente más alta que la de eucariotas. Los grupos bacterianos dominantes incluyeron Alphaproteobacteria, Gammaproteobacteria, Actinobacteria y Bacteroidia.
- Comunidades fúngicas: Los hongos representaron una parte importante de la riqueza eucariota, constituyendo aproximadamente un tercio de las ASVs en esta categoría. Entre ellos, se identificaron grupos conocidos por ser parásitos de microalgas, como Rozellomycota y Aphelidiomycota.
Identificando a los «buenos»: bacterias que promueven un cultivo saludable
El análisis permitió correlacionar la presencia de ciertos microorganismos con condiciones de cultivo «saludables» (definidas como una abundancia de D. armatus superior al 70%). En este grupo de «aliados» potenciales, destacaron varias bacterias:
- Geminocystis (ASV_2): Esta cianobacteria se asoció consistentemente con cultivos saludables en ambos tipos de reactores y durante los dos años de estudio. La presencia recurrente de la cianobacteria en condiciones óptimas y su asociación positiva en los análisis de redes la señalan como una potencial benefactora para D. armatus.
- Thiocapsa: Este género de Gammaproteobacteria se vinculó a condiciones saludables en el reactor de aguas residuales. Al ser una bacteria fotótrofa, podría beneficiar a Desmodesmus participando en el reciclaje de carbono, nitrógeno y fósforo.
- Ahniella (ASV_63): También una Gammaproteobacteria, mostró una interacción positiva con la microalga y apareció en condiciones saludables, especialmente en el tratamiento de aguas residuales, donde es una bacteria común en biopelículas microalgales.
- Bosea: Perteneciente a la clase Rhizobiales, se encontró en condiciones saludables en el reactor de fertilizantes y, en menor medida, en el de aguas residuales. Las bacterias de este grupo son comunes en la ficosfera (la zona que rodea a las algas) de cultivos industriales.
Detectando a los «malos»: patógenos bacterianos y fúngicos
De la misma manera, el estudio identificó microorganismos asociados a condiciones «no saludables» (con abundancia de D. armatus por debajo del 20%), revelando a los principales sospechosos de perjudicar el cultivo.
- Mycobacterium: Diversas variantes (ASVs) de esta bacteria aparecieron consistentemente en cultivos no saludables en ambos reactores y a lo largo del tiempo. La asociación negativa de Mycobacterium con D. armatus en los análisis de redes subraya su efecto potencialmente perjudicial, un hallazgo novedoso ya que su impacto en cultivos de microalgas no había sido documentado previamente.
- Flavobacterium: Aunque su papel puede ser complejo, ciertas variantes de esta bacteria se asociaron con condiciones no saludables y mostraron interacciones negativas. Algunas especies de Flavobacterium son conocidas por atacar a las microalgas, ya sea por contacto directo o liberando compuestos que degradan sus paredes celulares.
- Hongos parásitos: El estudio reafirmó el rol dañino de varios grupos de hongos y, por primera vez, identificó a otros como posibles amenazas para D. armatus:
- Paraphelidium tribonemae (ASV 8) y Aphelidium parallelum (ASV_39): Se identificaron por primera vez como posibles parásitos de D. armatus, al estar ligados a condiciones no saludables.
- Aphelidium desmodesmi (ASV_6) y Rhizophydium sp. (ASV 37): Confirmaron su conocido papel como patógenos, apareciendo en cultivos debilitados.
- Rozellomycota: Diferentes variantes de este grupo, conocidos parásitos de microalgas, se asociaron negativamente con la salud del cultivo en la mayoría de los casos.
Es interesante destacar que el rol de algunos hongos parece ser dependiente del contexto. Por ejemplo, una variante de Aphelidium desmodesmi (ASV_104) se asoció a condiciones no saludables en tres de los cuatro escenarios, pero a condiciones saludables en uno de ellos, lo que demuestra la complejidad de estas interacciones.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Implicaciones para el futuro de la producción de microalgas
Este estudio no solo mapea la compleja comunidad microbiana en los reactores de microalgas, sino que también proporciona información crucial para el manejo y la optimización de estos sistemas. La capacidad de identificar tempranamente un aumento en la población de patógenos como Mycobacterium o de hongos parásitos podría servir como una señal de alerta para prevenir el colapso de un cultivo.
Asimismo, comprender qué bacterias beneficiosas como Geminocystis prosperan junto a D. armatus abre la puerta al diseño de estrategias de «probióticos» para microalgas, donde se podrían inocular consorcios microbianos específicos para mejorar la resiliencia y la productividad del sistema.
En resumen, el camino hacia una producción de microalgas más robusta y eficiente pasa, inevitablemente, por comprender y gestionar el diminuto pero poderoso microbioma que las acompaña.
El estudio ha sido financiado por el Programa de Investigación e Innovación Horizonte 2020 de la Unión Europea a través del proyecto PRODIGIO: “Developing early-warning systems for improved microalgae PROduction and anaerobic DIGestION”, y por el proyecto INCEPTION “In-depth characterization of microalgal crop genomics for sustainable biomass production and circular bioeconomy” financiado por la Agencia Española de Investigación (AEI).
Contacto
Judith Traver-Azuara
Institute of Marine Sciences (ICM), CSIC
Barcelona 08003, Spain
Email: judithtraverazuara@icm.csic.es
Referencia (acceso abierto)
Traver-Azuara, J., Giner, C. R., García-Comas, C., Sánchez-Zurano, A., Ciardi, M., Acién, G., Bondarenko, S., Obiol, A., Massana, R., Sala, M. M., Logares, R., & Cermeño, P. (2025). Complex interplay between the microalgae and their microbiome in production raceways. Bioresource Technology, 432, 132650. https://doi.org/10.1016/j.biortech.2025.132650
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.