La industria mundial de la pesca y la acuicultura está en plena expansión. Con una producción total que alcanzó los 223.2 millones de toneladas en 2022 y un comercio que supera los 160 mil millones de dólares anuales, la demanda de proteína de alta calidad nunca ha sido tan alta. Para satisfacer esta demanda de forma sostenible, la acuicultura moderna necesita optimizar la eficiencia alimenticia, mejorar la reproducción, regular el crecimiento y descifrar los mecanismos de resistencia a enfermedades.
La clave para lograr estos avances se encuentra en los datos. Específicamente, en los datos «multi-ómicos»: la información combinada de la genómica, transcriptómica, epigenética y proteómica de un organismo. Sin embargo, hasta ahora, estos datos vitales para las especies de peces estaban fragmentados, dispersos en diversas publicaciones o bases de datos incompletas.
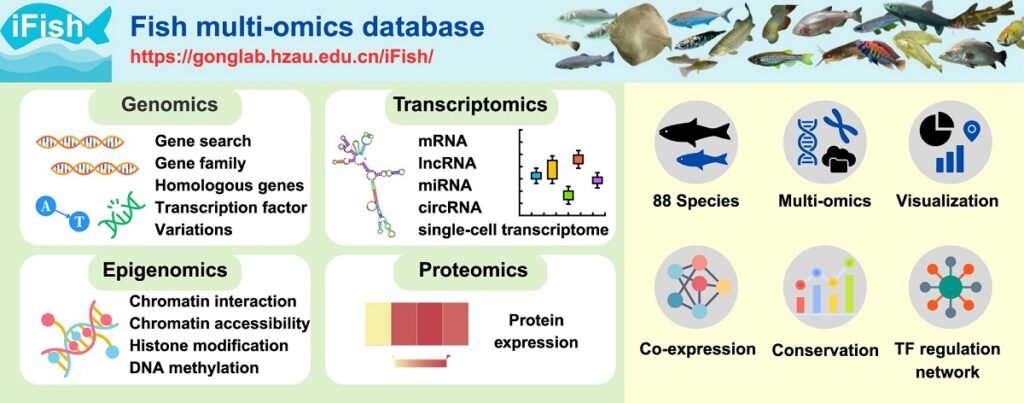
Para solucionar este problema, un equipo de investigadores de la Huazhong Agricultural University, ha desarrollado iFish, una plataforma integral diseñada para ser el recurso multi-ómico más completo para la investigación piscícola hasta la fecha.
- 1 Conclusiones clave
- 2 El desafío de los datos ómicos en la acuicultura
- 3 iFish: una solución integral para la investigación piscícola
- 4 ¿Qué contiene la base de datos iFish?
- 5 El poder del ARN no codificante (ncRNA): una ventaja clave de iFish
- 6 Más que solo datos: herramientas y funcionalidades de iFish
- 7 Un caso de estudio: explorando la determinación sexual en pez cebra
- 8 Implicaciones para el futuro de la acuicultura
- 9 Conclusión: el futuro es multi-ómico
- 10 Entradas relacionadas:
Conclusiones clave
- Se ha desarrollado iFish, la plataforma más completa hasta la fecha, que integra datos multi-ómicos (genoma, transcriptoma, epigenoma y proteoma) de 88 especies de peces.
- La base de datos centraliza una cantidad masiva de información, incluyendo 137 millones de polimorfismos de nucleótido único (SNPs), 2.7 millones de anotaciones de genes y 192,107 factores de transcripción.
- Una fortaleza distintiva de iFish es su rico repositorio de ARN no codificantes (ncRNAs), como lncRNAs y miRNAs, cuya función es crucial para la biología de los peces pero estaba poco caracterizada.
- La plataforma ofrece herramientas fáciles de usar para visualización, descarga y análisis funcional, incluyendo redes de coexpresión génica y de regulación de factores de transcripción.
- iFish está diseñada para acelerar la investigación genética y la mejora genética en la acuicultura, facilitando el descubrimiento de genes ligados al crecimiento, la resistencia a enfermedades y la determinación del sexo.
El desafío de los datos ómicos en la acuicultura
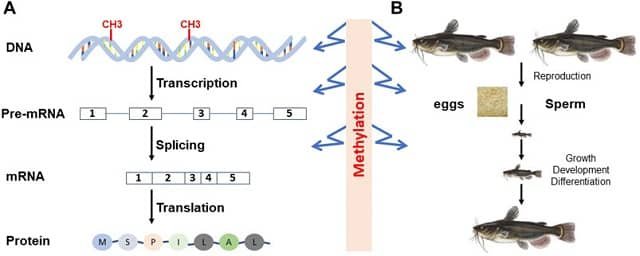
Los peces son el grupo más diverso de vertebrados y juegan un papel fundamental en la nutrición global y el equilibrio ecológico. Investigar su biología para mejorar la acuicultura requiere una comprensión profunda de su arquitectura genómica, sus redes de regulación génica y sus mecanismos epigenéticos.
En los últimos años, los científicos han utilizado diferentes enfoques ómicos para estudiar, por ejemplo, los marcadores genéticos (SNPs) relacionados con el crecimiento en el bagre amarillo o cómo la metilación del ADN influye en los espermatozoides X e Y.
Sin embargo, las bases de datos existentes eran limitadas. Algunas se centraban solo en el pez cebra, otras cubrían pocas especies o se especializaban únicamente en un tipo de datos, como los microARNs (miRNA) o los elementos transponibles. Faltaba una plataforma que integrara todo.
Según los autores del estudio, las bases de datos anteriores no eran «suficientemente completas» en cuanto a especies y tipos de datos. Aspectos cruciales como la variación genética, la identificación de ARN no codificantes (ncRNAs) y los conjuntos de datos epigenómicos eran insuficientes para la mayoría de las especies.
iFish: una solución integral para la investigación piscícola
Para llenar este vacío, se creó iFish (accesible en https://gonglab.hzau.edu.cn/iFish/), una base de datos rigurosamente seleccionada que integra múltiples capas de información ómica para 88 especies de peces.
Esta colección masiva de datos, que supera los 40 TB, incluye tanto especies modelo (como el pez cebra) como especies de gran importancia económica, entre ellas:
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
- Salmón del Atlántico (Salmo salar)
- Bacalao del Atlántico (Gadus morhua)
- Trucha arcoíris (Oncorhynchus mykiss)
- Dorada (Sparus aurata)
- Carpa herbívora (Ctenopharyngodon idella)
- Lubina de boca grande (Micropterus salmoides)
- Bagre amarillo (Pelteobagrus fulvidraco)
iFish no es solo un almacén; es una plataforma de análisis que permite a los usuarios navegar, visualizar y descargar datos de forma interactiva.
¿Qué contiene la base de datos iFish?
La plataforma iFish está organizada en módulos que cubren cuatro áreas principales de la biología molecular, procesando un total de 13,377 muestras de secuenciación:
Genómica (El «mapa» genético)
Este módulo proporciona la base del ADN. Integra 88 ensamblajes genómicos y datos de 884 proyectos de secuenciación del genoma completo (WGS). A partir de esto, los investigadores identificaron:
- 137.04 millones de SNPs (polimorfismos de nucleótido único).
- 45.52 millones de InDels (inserciones/deleciones).
- 2.7 millones de entradas de anotación de genes.
- 192,107 factores de transcripción (TFs), que son las proteínas que «encienden» o «apagan» los genes.
- Análisis de genes homólogos entre especies.
Transcriptómica (Qué genes se están «usando»)
Aquí se analiza la expresión génica (ARN). iFish procesó 9,797 conjuntos de datos de ARN-seq a granel, 293 de ARN-seq de célula única (scRNA-seq) y 840 de microARN-seq. Esto permitió cuantificar:
- 1.87 millones de ARNm (ARN mensajeros).
- 287,873 lncRNAs (ARN largos no codificantes).
- 197,554 circRNAs (ARN circulares).
- 6,068 miRNAs (microARNs) maduros.
Epigenómica (Cómo se regula el «mapa»)
La epigenética estudia los interruptores químicos que, sin cambiar el ADN, controlan qué genes se activan. iFish integra 1,563 conjuntos de datos epigenéticos para mapear:
- Modificaciones de histonas.
- Accesibilidad de la cromatina (qué partes del ADN están «abiertas» para su uso).
- Metilación del ADN.
- Interacciones tridimensionales de la cromatina (cómo se organiza el ADN en el espacio).
Proteómica (Las «máquinas» funcionales)
Finalmente, la proteómica analiza las proteínas, los productos finales de los genes. Este módulo incluye 50 proyectos para perfilar la expresión de proteínas.
El poder del ARN no codificante (ncRNA): una ventaja clave de iFish
Una de las fortalezas que distinguen a iFish es su rico repositorio de información sobre ARN no codificantes (ncRNAs).
En los últimos años, los científicos han descubierto que los ncRNAs, que antes se consideraban «ADN basura», son en realidad reguladores biológicos cruciales. En los peces, se ha demostrado que los ncRNAs desempeñan funciones vitales en el desarrollo y en la respuesta inmune. Por ejemplo, el estudio menciona cómo un lncRNA específico (AANCR) promueve respuestas antivirales, y cómo un miR-722 ayuda a modular la respuesta inmune contra bacterias en el lenguado.
A pesar de su importancia, los perfiles de expresión y las funciones de los ncRNAs en peces estaban poco caracterizados, y faltaba una plataforma completa. iFish aborda esto sistemáticamente, identificando y caracterizando el panorama de expresión de lncRNAs, circRNAs y miRNAs, e incluso investigando su conservación entre diferentes especies de peces.
Más que solo datos: herramientas y funcionalidades de iFish
El verdadero poder de iFish no reside solo en la cantidad de datos, sino en cómo permite a los investigadores usarlos. La plataforma ofrece herramientas de análisis funcional que convierten números brutos en conocimiento biológico.
Dos de las herramientas más potentes son:
- Redes de coexpresión génica: iFish ha calculado 289 millones de pares de genes que se «encienden» y «apagan» juntos en 19 especies. Esto permite a un investigador buscar un gen de interés y descubrir instantáneamente con qué otros genes está colaborando.
- Redes de regulación de TF: La base de datos mapea 281 millones de pares regulatorios entre factores de transcripción (TFs) y sus genes objetivo en 46 especies. Esto ayuda a descifrar cómo se controlan las redes genéticas.
Un caso de estudio: explorando la determinación sexual en pez cebra
Para demostrar la utilidad de iFish, los investigadores realizaron un caso de estudio sobre el gen dmrt1 en el pez cebra.
La determinación del sexo es crucial en la acuicultura, ya que el cultivo de poblaciones de un solo sexo (monosexo) puede ser muy valioso. El gen dmrt1 es conocido por ser un actor clave en el desarrollo sexual masculino en muchos peces.
Al usar iFish, los investigadores:
- Buscaron el gen «dmrt1» en el módulo de búsqueda de genes.
- La plataforma mostró la información del gen, su ubicación en el genoma (visualizada con JBrowse) y su perfil de expresión en diferentes tejidos.
- Tal como se esperaba, los resultados mostraron una alta expresión de dmrt1 específicamente en el tejido testicular, confirmando los informes previos y validando la precisión de la base de datos.
Además, pudieron usar la herramienta de «Función» para generar instantáneamente una red de coexpresión, mostrando todos los genes que trabajan junto con dmrt1 en el testículo del pez cebra.
Implicaciones para el futuro de la acuicultura
iFish representa un salto adelante significativo para la investigación piscícola. Al unificar datos genómicos, transcriptómicos, epigenéticos y proteómicos bajo una misma plataforma accesible, reduce drásticamente el tiempo y el esfuerzo necesarios para realizar análisis complejos.
Para los profesionales de la acuicultura y los genetistas, esto tiene implicaciones prácticas directas:
- Mejora genética acelerada: Los criadores pueden usar iFish para identificar más rápidamente marcadores genéticos (SNPs) y genes asociados con rasgos deseables, como un crecimiento más rápido o una mejor conversión alimenticia.
- Salud y enfermedad: Los investigadores pueden explorar los perfiles de expresión génica (como los ncRNAs) y los cambios epigenéticos (como la metilación del ADN) que ocurren cuando un pez responde a un patógeno o al estrés ambiental.
- Optimización reproductiva: La capacidad de investigar redes de determinación sexual (como el ejemplo de dmrt1) facilita el desarrollo de poblaciones monosexo.
- Investigación evolutiva: La plataforma permite análisis de conservación entre especies, ayudando a entender la evolución de los peces.
Conclusión: el futuro es multi-ómico
La base de datos iFish es, hasta la fecha, la plataforma más completa que integra, analiza y almacena sistemáticamente datos multi-ómicos para peces. Al proporcionar una «ventanilla única» fácil de usar, los creadores esperan que iFish sirva como un recurso fundamental para avanzar en las investigaciones genéticas, de regulación transcripcional y epigenéticas.
Para una industria acuícola que depende de la innovación científica para satisfacer la creciente demanda mundial de proteínas, herramientas como iFish no son solo un avance académico; son motores esenciales para la mejora genética y las aplicaciones prácticas que definirán el futuro sostenible del sector.
Contacto
Ze-Xia Gao
1 Hubei Hongshan Laboratory, College of Informatics, Huazhong Agricultural University
Wuhan 430070, China
Email: gaozx@mail.hzau.edu.cn
Referencia (acceso abierto)
Ruoyan Liu, Qin Tang, Peng Sun, Weiwei Jin, Yanbo Yang, Xiaohong Wu, Wen Cao, Xiaohui Niu, Jing Gong, Ze-Xia Gao, iFish: a comprehensive multi-omics database for fish genetics, transcriptional regulation, and epigenetic research, Nucleic Acids Research, 2025;, gkaf1026, https://doi.org/10.1093/nar/gkaf1026
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.