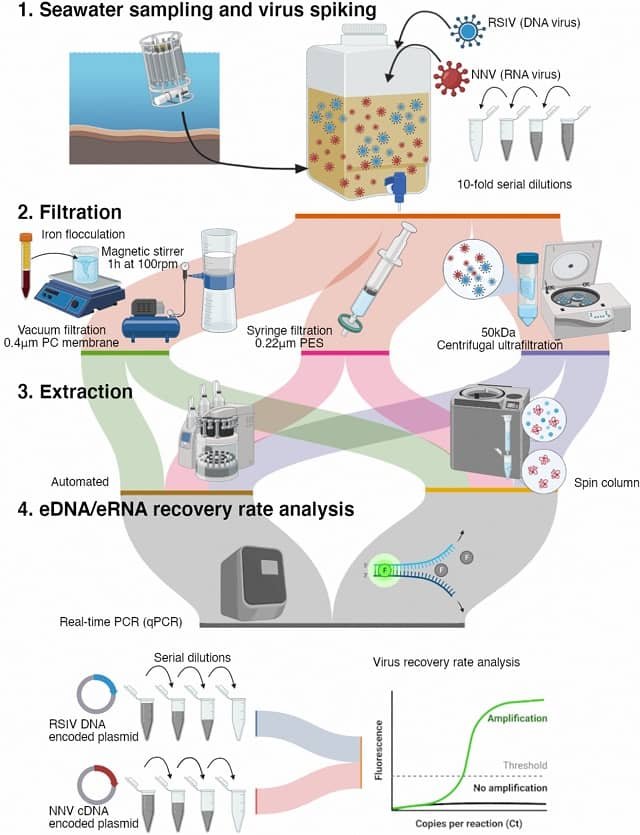
En la última década, el ADN ambiental (eDNA) ha revolucionado el monitoreo de la biodiversidad. Al filtrar una simple muestra de agua, podemos identificar qué especies habitan un ecosistema, desde bacterias hasta vertebrados, detectando incluso organismos crípticos, invasores o en peligro de extinción. Esta herramienta ha potenciado enormemente la evaluación de pesquerías y la monitorización de la biodiversidad.
Sin embargo, el eDNA tiene una limitación fundamental: nos dice quién está presente, pero muy poco sobre qué está haciendo. Inferir actividades fisiológicas dinámicas, como la reproducción, el estrés o el estado de salud, sigue siendo un desafío.
Aquí es donde el ARN ambiental (eRNA) está comenzando a cambiar el paradigma. A diferencia del ADN, el ARN es un indicador de la actividad génica en tiempo real. Un nuevo estudio realizado por científicos de la The University of Tokyo explora cómo el eRNA puede usarse específicamente para inferir uno de los comportamientos más cruciales y difíciles de observar en el medio acuático: el desove.
- 1 Conclusiones clave
- 2 ¿Por qué el ARN ambiental (eRNA)?
- 3 Buscando los marcadores perfectos
- 4 Una correlación directa entre eRNA y comportamiento
- 5 Validación por video: el eRNA coincide con el comportamiento
- 6 Implicaciones para la acuicultura y la gestión pesquera
- 7 Conclusión
- 8 Entradas relacionadas:
Conclusiones clave
- El ADN ambiental (eDNA) es excelente para saber qué especies están presentes, pero no para inferir sus actividades fisiológicas dinámicas, como la reproducción.
- El ARN ambiental (eRNA) emerge como una herramienta que sí puede capturar estas actividades biológicas dinámicas, ya que el ARN es específico de los tejidos (como gametos o moco) y refleja el estado funcional del organismo.
- El estudio identificó el gen klhl10, específico del testículo, como un marcador de eRNA sensible. Sus niveles en el agua aumentaron significativamente solo cuando se produjo el apareamiento de los peces.
- Se encontró una correlación positiva directa entre la duración de los intentos de apareamiento (grabados en video) y el aumento de los niveles de klhl10 en el agua, validando su uso para inferir el desove.
- Esta técnica beneficia a la acuicultura (monitoreo reproductivo), la evaluación ambiental y la conservación, especialmente para localizar zonas de desove de especies pelágicas difíciles de observar.
¿Por qué el ARN ambiental (eRNA)?
Mientras que el eDNA es relativamente estable, el ARN es más dinámico y, crucialmente, es específico de cada tejido en organismos multicelulares. En los peces, las principales fuentes de eRNA en el agua provienen del moco epitelial (piel y branquias) y de los gametos (esperma y óvulos) liberados durante la reproducción.
El gran desafío para los gestores pesqueros ha sido localizar las zonas de desove naturales de especies pelágicas clave, como sardinas y anchoas, cuyos stocks fluctúan enormemente. Si se liberan grandes cantidades de gametos al medio ambiente durante el desove, los marcadores de eRNA específicos de esos gametos deberían aumentar drásticamente en el agua, actuando como una «baliza» científica que indica que el desove está ocurriendo.
Buscando los marcadores perfectos
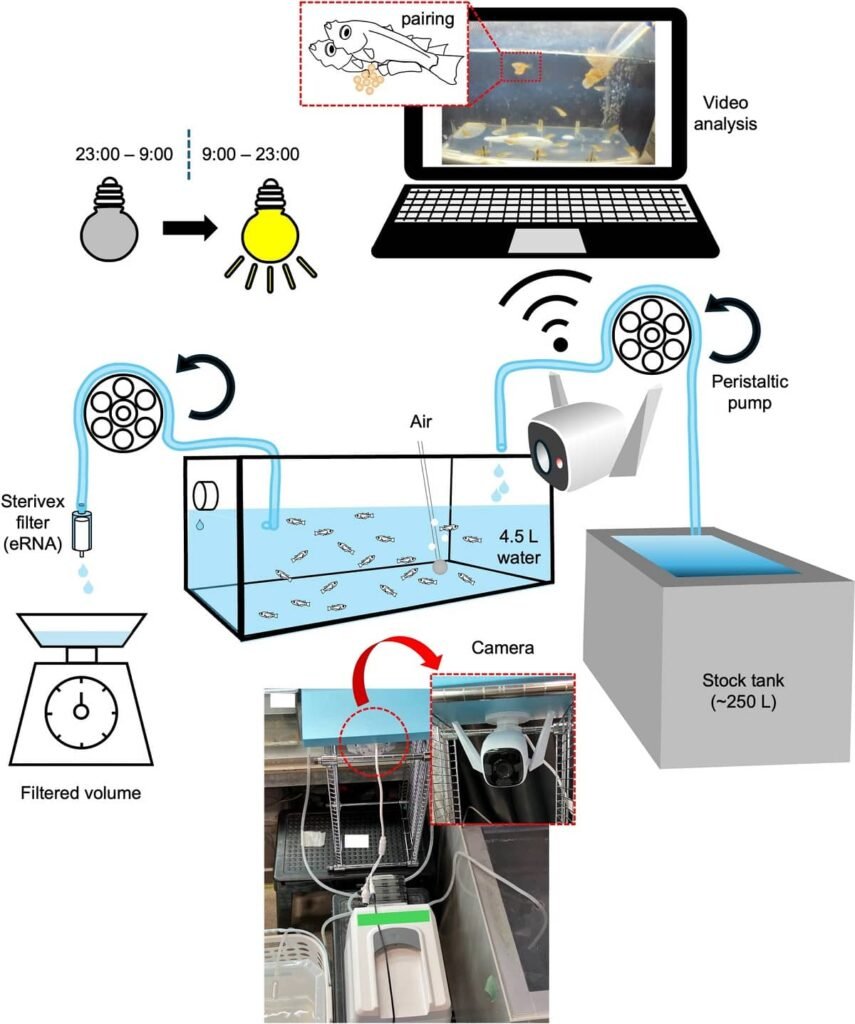
Para probar esta hipótesis, los investigadores necesitaban encontrar los genes correctos. Utilizaron al medaka (una especie de pez modelo) porque su comportamiento reproductivo es bien conocido: desovan diariamente por la mañana bajo condiciones específicas.
El equipo buscó dos tipos de marcadores:
- Un marcador de desove: Un gen que se exprese exclusivamente en las gonadas (testículos u ovarios).
- Un marcador de control (epitelial): Un gen expresado constantemente en la piel o las branquias, para confirmar la presencia de peces independientemente del desove.
Tras analizar la literatura y datos de transcriptómica, seleccionaron dos candidatos principales:
- Marcador de desove: klhl10. Este gen demostró ser altamente específico de los testículos, estando involucrado en la formación de espermátidas.
- Marcador de control: muc5ac. Este gen de mucina se expresa en la piel y las branquias, pero no en las gónadas, sirviendo como un indicador epitelial.
El experimento fue directo: colocaron diferentes combinaciones de medaka en tanques (solo machos, solo hembras y parejas mixtas). Luego, filtraron el agua de los tanques donde se observó apareamiento y de los tanques donde no ocurrió, y midieron los niveles de eRNA de klhl10 y muc5ac usando qPCR.
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Una correlación directa entre eRNA y comportamiento
Los resultados fueron claros y contundentes, demostrando que el eRNA es un indicador sensible de la actividad reproductiva.
El marcador klhl10 solo aparece durante el apareamiento
El hallazgo más importante fue que los niveles del marcador de desove klhl10 fueron significativamente más altos en el agua de los tanques donde las parejas de medaka se aparearon.
En cambio, en los tanques que contenían solo machos, solo hembras o parejas que no se aparearon, los niveles de klhl10 fueron en su mayoría indetectables (ver Figura 2A del estudio). Esto confirma que la presencia de este eRNA en el agua está directamente causada por la liberación de gametos durante el apareamiento.
El marcador muc5ac es constante
Por el contrario, el marcador de control epitelial muc5ac (moco) fue detectable en todas las muestras de agua donde había peces. Esto era de esperar, ya que los peces liberan moco constantemente.
Crucialmente, los niveles de muc5ac no mostraron diferencias entre los grupos que se aparearon y los que no. Esto demuestra que el acto de aparearse no aumenta la liberación de moco epitelial, reforzando que klhl10 es un marcador específico para el desove y no para un aumento general de la actividad.
Validación por video: el eRNA coincide con el comportamiento
Para confirmar la relación temporal, el equipo realizó un experimento más detallado. Utilizaron un «tanque aislado» y grabaron continuamente el comportamiento de los peces con una cámara infrarroja, mientras tomaban muestras de agua cada hora.
Los datos mostraron que el aumento en los transcritos de klhl10 en el agua coincidió perfectamente con la duración de los intentos de apareamiento observados en el video. Un análisis de correlación de Pearson confirmó una correlación positiva significativa (r = 0.4776; p < 0.001) entre la duración del apareamiento y los niveles de klhl10.
Además, el eRNA demostró ser un indicador de «tiempo real». Los niveles de klhl10 solo fueron detectables en la mañana (cuando el medaka desova) y fueron indetectables durante el resto del día. Esto se debe a que el ARN se degrada rápidamente en el agua (el estudio estimó la vida media de muc5ac en solo 15 minutos a 25°C), lo cual es una ventaja: asegura que la señal detectada corresponde a un evento reciente.
Implicaciones para la acuicultura y la gestión pesquera
Los hallazgos de este estudio tienen aplicaciones prácticas directas para el sector. El desarrollo de un marcador de eRNA basado en qPCR, sensible y específico para el desove, es altamente valioso para el trabajo de campo.
- Gestión de pesquerías: La principal aplicación es la localización de zonas de desove de especies pelágicas. Actualmente, se debate sobre dónde desovan especies como la caballa o el paparda del Pacífico. Mientras que el eDNA solo puede rastrear la migración de estos peces, la adición de evidencia de eRNA (como klhl10) podría proporcionar una imagen concluyente de dónde y cuándo están desovando. Esta tecnología es relativamente de bajo costo y los resultados pueden obtenerse en horas, incluso a bordo de buques de investigación.
- Acuicultura: El monitoreo del eRNA puede beneficiar directamente a la acuicultura. En lugar de métodos invasivos o de observación directa que pueden estresar a los reproductores, los productores podrían tomar muestras de agua de los tanques de reproductores para confirmar el éxito y el momento del desove de forma no invasiva.
- Evaluación ambiental y conservación: Esta herramienta permite monitorear las actividades fisiológicas de las especies en el campo. Además del desove, los investigadores sugieren que otros marcadores de eRNA (como el muc5ac) podrían investigarse como indicadores de estrés, enfermedades o niveles de actividad en los peces.
Conclusión
Este estudio cierra una brecha crítica que el eDNA no podía cubrir. Al pasar de simplemente identificar la presencia de una especie a detectar sus actividades fisiológicas dinámicas, el ARN ambiental ofrece una visión mucho más rica de lo que ocurre bajo el agua.
La identificación de klhl10 como un marcador de eRNA sensible y específico para el desove proporciona una herramienta poderosa y práctica. Permite a los científicos y gestores «escuchar» las conversaciones moleculares de la reproducción de los peces, un avance que sin duda beneficiará la sostenibilidad de la acuicultura, la evaluación ambiental y la conservación de la biodiversidad marina.
Contacto
Marty Kwok-Shing Wong
Laboratory of Physiology, Atmosphere and Ocean Research Institute, The University of Tokyo
5-1-5 Kashiwanoha, Kashiwa City, 277-8564, Chiba, Japan
Email: martywong@aori.u-tokyo.ac.jp
Referencia (acceso abierto)
Aminaka, Y., Wong, M. K., Yada, T., & Hyodo, S. (2025). The use of environmental RNA for inferring fish spawning behavior. Scientific Reports, 15(1), 1-14. https://doi.org/10.1038/s41598-025-23861-8
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.