Edimburgo, Reino Unido.- El metabarcoding del ADN ambiental es relativamente una nueva herramienta de monitoreo con un creciente número de aplicaciones, como la fácil detección de especies en muestras ambientales. Un estudio concluye que el ADN ambiental ofrece un potencial significativo para monitorear las especies peligrosas para la acuicultura.
La industria de la acuicultura de salmónidos viene siendo afectados por enfermedades parasitarias y frecuentemente está sometida a mortalidades masivas de los peces de cultivo debido a las toxinas producidas por las floraciones algales peligrosas.
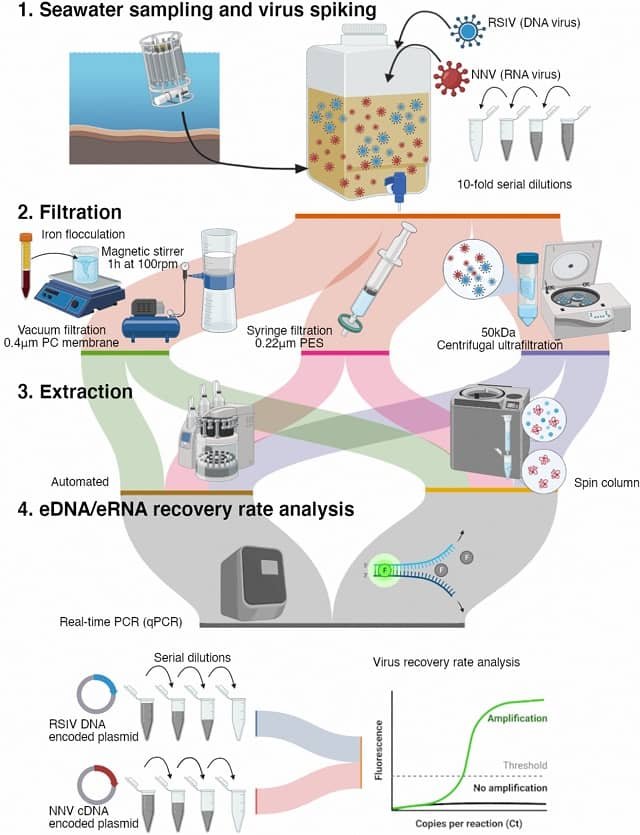
El análisis del ADN ambiental (ADNa) es un enfoque molecular emergente para la identificación de especies de muestras que contienen ADN celular y ADN extracelular desprendidos de todos los organismos vivientes. El ADNa ha sido empleado con éxito para detectar y monitorear comunidades y poblaciones microbianas, y es una herramienta útil para los sistemas de monitoreo temprano debido a que permite la detección de especies que son crípticas, inaccesibles y de baja abundancia.
Los métodos de secuenciamiento de próxima generación vienen siendo usados para identificar los organismos acuáticos en base a las muestras de ADNa. El metabarcoding es la técnica frecuentemente más empleado.
Aunque la detección de los patógenos es importante para la industria de la acuicultura, también es importante evaluar si la abundancia de los organismos patogénicos han excedido los umbrales establecidos con respecto a la seguridad del alimento o la calidad del agua.
Los investigadores de la University of Edinburgh, University of Glasgow, Instituto Oswaldo Cruz (Brasil), University of Tromsø (Noruega) y Ferskvannsbiologen, Ltd. (Noruega) implementaron un estudio para establecer si los patógenos eucariotas y las algas peligrosas pueden ser detectados e identificados usando el enfoque del metabarcoding.
De acuerdo con los resultados de los científicos, el metabarcoding ADNa de la región 18S SSU rDNA v9 puede identificar múltiples patógenos en la acuicultura, de la mezcla compleja de organismos presentes en el agua de mar. “Además, nuestras comparaciones indican que la detección puede ser alcanzada usando la plataforma de secuenciamiento de bajo costo Ion Torrent” dijeron.
“Nosotros fuimos capaces de identificar dos parásitos (Lepeophtheirus salmonis y Paramoeba perurans) a nivel de especies, mientras que las especies de microalgas Prymnesium parvum, Pseudo-nitzschia seriata, y P. delicatissima pueden ser asignados correctamente solo a nivel de género” reportan los científicos. {mprestriction ids=»*»}
Mantente siempre informado
Únete a nuestras comunidades para recibir al instante las noticias, informes y análisis más importantes del sector acuícola.
Ellos manifiestan que Illumina y Ion Torrent proveen resultados casi idénticos en términos de composición de comunidad en las muestras, mientras que Ion Torrent fue más sensible en detectar la riqueza de especies.
“Nuestro hallazgo indica que el metabarcoding del ADNa ofrece un potencial significativo en el monitoreo de especies peligrosas para la acuicultura, y para este propósito, el secuenciamiento de bajo costo Ion Torrent es más exacto que Illumina en determinar las diferencias en la abundancia relativa entre las muestras.
Referencia (abierto):
Lucy Peters, Sofie Spatharis, Maria Augusta Dario, Toni Dwyer, Inaki J. T. Roca, Anna Kintner, Øyvind Kanstad-Hanssen, Martin S. Llewellyn and Kim Praebel. Environmental DNA: A New Low-Cost Monitoring Tool for Pathogens in Salmonid Aquaculture. Front. Microbiol., 07 December 2018 | https://doi.org/10.3389/fmicb.2018.03009
https://www.frontiersin.org/articles/10.3389/fmicb.2018.03009/full {/mprestriction}
Editor de la revista digital AquaHoy. Biólogo Acuicultor titulado por la Universidad Nacional del Santa (UNS) y Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomados en Innovación Empresarial y Gestión de la Innovación. Posee amplia experiencia en el sector acuícola y pesquero, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA). Ha sido consultor senior en vigilancia tecnológica, formulador y asesor de proyectos de innovación, y docente en la UNS. Es miembro del Colegio de Biólogos del Perú y ha sido reconocido por la World Aquaculture Society (WAS) en 2016 por su aporte a la acuicultura.